备注
Go to the end 下载完整的示例代码。或者通过浏览器中的MysterLite或Binder运行此示例
主成分回归与偏最小平方回归#
该实施例比较 Principal Component Regression (PCR)和 Partial Least Squares Regression (PLS)在玩具数据集上。我们的目标是说明当目标与数据中具有低方差的一些方向强相关时,SCS如何优于PCR。
PCR是由两个步骤组成的回归器:首先, PCA 应用于训练数据,可能执行降维;然后,在变换后的样本上训练回归器(例如线性回归器)。在 PCA ,转换完全是无监督的,这意味着不使用有关目标的信息。因此,PCR在一些目标与之强相关的数据集中可能表现不佳 directions 方差较低的。事实上,PCA的降维将数据投影到较低维空间中,其中投影数据的方差沿着每个轴被贪婪地最大化。尽管它们对目标具有最强的预测能力,但方差较低的方向将被丢弃,并且最终的回归量将无法利用它们。
最大限度地提高了效率,也是一个回归量,它与PCR非常相似:在对转换后的数据应用线性回归量之前,它还对样本进行了降维。与PCR的主要区别在于,偏头痛转换是受监督的。因此,正如我们在这个例子中看到的那样,它不会受到我们刚才提到的问题的影响。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
数据#
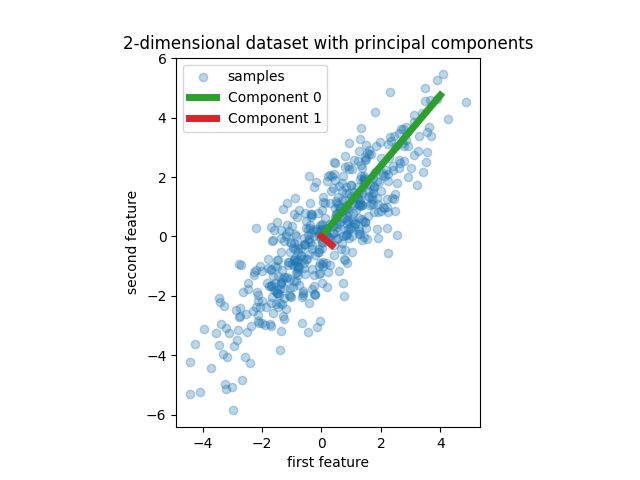
我们首先创建一个具有两个特征的简单数据集。在我们深入研究PCR和SPL之前,我们先适应PCA估计器来显示该数据集的两个主要成分,即解释数据中最大方差的两个方向。
import matplotlib.pyplot as plt
import numpy as np
from sklearn.decomposition import PCA
rng = np.random.RandomState(0)
n_samples = 500
cov = [[3, 3], [3, 4]]
X = rng.multivariate_normal(mean=[0, 0], cov=cov, size=n_samples)
pca = PCA(n_components=2).fit(X)
plt.scatter(X[:, 0], X[:, 1], alpha=0.3, label="samples")
for i, (comp, var) in enumerate(zip(pca.components_, pca.explained_variance_)):
comp = comp * var # scale component by its variance explanation power
plt.plot(
[0, comp[0]],
[0, comp[1]],
label=f"Component {i}",
linewidth=5,
color=f"C{i + 2}",
)
plt.gca().set(
aspect="equal",
title="2-dimensional dataset with principal components",
xlabel="first feature",
ylabel="second feature",
)
plt.legend()
plt.show()
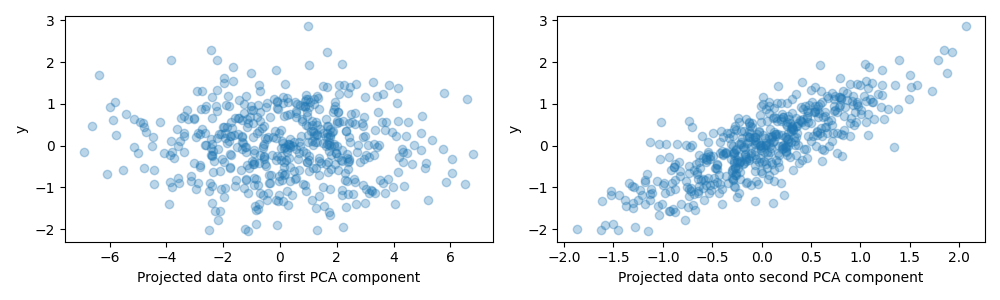
出于本示例的目的,我们现在定义目标 y 使得它与具有小方差的方向强相关。为此,我们将 X 将其添加到第二个组件上,并为其添加一些噪音。
y = X.dot(pca.components_[1]) + rng.normal(size=n_samples) / 2
fig, axes = plt.subplots(1, 2, figsize=(10, 3))
axes[0].scatter(X.dot(pca.components_[0]), y, alpha=0.3)
axes[0].set(xlabel="Projected data onto first PCA component", ylabel="y")
axes[1].scatter(X.dot(pca.components_[1]), y, alpha=0.3)
axes[1].set(xlabel="Projected data onto second PCA component", ylabel="y")
plt.tight_layout()
plt.show()
对一个组成部分的预测和预测能力#
我们现在创建两个回归器:PCR和SCS,为了说明的目的,我们将组件数量设置为1。在将数据提供给PCR的PCA步骤之前,我们首先按照良好实践的建议对其进行标准化。最大化估计器具有内置的扩展功能。
对于这两个模型,我们将投影数据绘制到针对目标的第一个组件上。在这两种情况下,该预测数据都是回归器将用作训练数据的数据。
from sklearn.cross_decomposition import PLSRegression
from sklearn.decomposition import PCA
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import train_test_split
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=rng)
pcr = make_pipeline(StandardScaler(), PCA(n_components=1), LinearRegression())
pcr.fit(X_train, y_train)
pca = pcr.named_steps["pca"] # retrieve the PCA step of the pipeline
pls = PLSRegression(n_components=1)
pls.fit(X_train, y_train)
fig, axes = plt.subplots(1, 2, figsize=(10, 3))
axes[0].scatter(pca.transform(X_test), y_test, alpha=0.3, label="ground truth")
axes[0].scatter(
pca.transform(X_test), pcr.predict(X_test), alpha=0.3, label="predictions"
)
axes[0].set(
xlabel="Projected data onto first PCA component", ylabel="y", title="PCR / PCA"
)
axes[0].legend()
axes[1].scatter(pls.transform(X_test), y_test, alpha=0.3, label="ground truth")
axes[1].scatter(
pls.transform(X_test), pls.predict(X_test), alpha=0.3, label="predictions"
)
axes[1].set(xlabel="Projected data onto first PLS component", ylabel="y", title="PLS")
axes[1].legend()
plt.tight_layout()
plt.show()
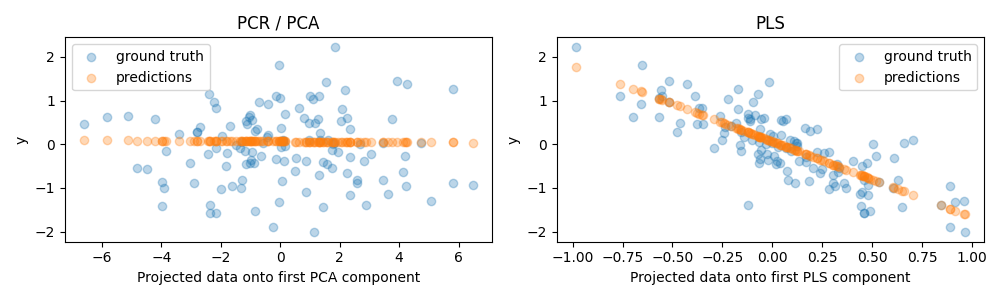
正如预期的那样,PCR的无监督PCA转换已经删除了第二个分量,即方差最低的方向,尽管它是最具预测性的方向。这是因为PCA是一种完全无监督的转换,导致投影数据对目标的预测能力较低。
另一方面,由于在转换过程中使用目标信息,最大化回归器能够捕获方差最低的方向的影响:它可以识别出这个方向实际上是最具预测性的。我们注意到,第一个最大化方差分量与目标负相关,这是因为特征量的符号是任意的。
我们还打印了两个估计器的R平方分数,这进一步证实了在这种情况下,在这种情况下,SCS是比PCR更好的替代方案。负R平方表明PCR的表现比简单预测目标平均值的回归量差。
print(f"PCR r-squared {pcr.score(X_test, y_test):.3f}")
print(f"PLS r-squared {pls.score(X_test, y_test):.3f}")
PCR r-squared -0.026
PLS r-squared 0.658
最后一点,我们注意到具有2个成分的PCR的性能与最大限度地好:这是因为在这种情况下,PCR能够利用对目标具有最强捕食力的第二个成分。
pca_2 = make_pipeline(PCA(n_components=2), LinearRegression())
pca_2.fit(X_train, y_train)
print(f"PCR r-squared with 2 components {pca_2.score(X_test, y_test):.3f}")
PCR r-squared with 2 components 0.673
Total running time of the script: (0分0.433秒)
相关实例
Gallery generated by Sphinx-Gallery <https://sphinx-gallery.github.io> _