定义新模型类#
本文档描述如何向包中添加模型或创建用户定义的模型。简而言之,我们需要定义所有的模型参数并编写一个计算模型的函数,也就是说,计算实现模型的数学函数。如果模型是可拟合的,如果要使用线性拟合算法,则需要一个计算参数导数的函数;如果要使用非线性拟合器,则需要一个可选函数。
基本定制型号#
大多数情况下 custom_model decorator提供了一种简单的方法来制作新的 Model 从现有的Python可调用文件初始化。以下示例演示如何设置由两个高斯函数组成的模型:
import numpy as np
import matplotlib.pyplot as plt
from astropy.modeling.models import custom_model
from astropy.modeling.fitting import TRFLSQFitter
# Define model
@custom_model
def sum_of_gaussians(x, amplitude1=1.0, mean1=-1.0, sigma1=1.0,
amplitude2=1.0, mean2=1.5, sigma2=1.0):
return (amplitude1 * np.exp(-0.5 * ((x - mean1) / sigma1)**2) +
amplitude2 * np.exp(-0.5 * ((x - mean2) / sigma2)**2))
# Generate fake data with some noise
rng = np.random.default_rng(0)
x = np.linspace(-5., 5., 200)
m_ref = sum_of_gaussians(amplitude1=2., mean1=-0.5, sigma1=0.4,
amplitude2=0.5, mean2=2., sigma2=1.0)
y = m_ref(x) + rng.normal(0., 0.05, x.shape)
# Fit model to data
m_init = sum_of_gaussians()
fit = TRFLSQFitter()
m = fit(m_init, x, y)
# Plot the data and the best fit
fig, ax = plt.subplots()
ax.plot(x, y, 'o', color='k')
ax.plot(x, m(x))
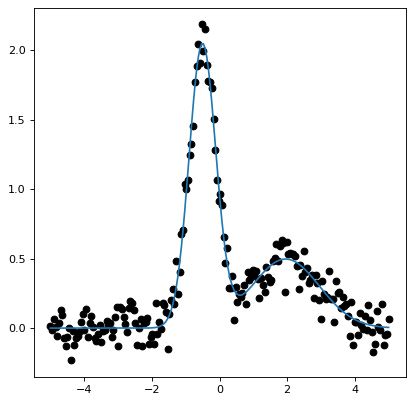
此修饰器还支持设置模型的 fit_deriv 以及创建具有多个输入的模型。请注意,从具有多个输出的函数创建模型时,关键字参数 n_outputs 必须设置为函数的输出数。它也可以用作正常的工厂功能(例如 SumOfGaussians = custom_model(sum_of_gaussians) ),而不是作为一名装饰师。请参阅 custom_model 有关更多示例的文档。
一维高斯模型的逐步定义#
中描述的示例 Basic custom models 可以用于大多数简单的情况,但是下面的部分描述了如何构造一般的模型类。定义一个完整的模型类可能是可取的,例如,提供更专业的参数,或者实现basic不支持的特殊功能 custom_model 工厂功能。
下面以1-D高斯模型为例解释详细信息。模型有两个基类。如果模型适合,则应继承 FittableModel ;如果不是的话,它应该是子类 Model .
如果模型采用参数,则应在模型的类定义中使用 Parameter 描述符。参数构造函数的所有参数都是可选的,并且可以包括该参数的默认值、参数的文本说明(用于 help 和文档生成),以及参数值的缺省约束和定制的获取/设置方法。还可以为每个参数定义一个“验证器”方法,使定制代码能够根据模型定义检查该参数的值是否有效(例如,它是否必须为非负数)。参见中的示例 Parameter.validator 了解更多详细信息。请注意,如果酸洗模型很重要,则应将验证器函数直接分配给实例 Parameter._validator 而不是使用装饰物。
from astropy.modeling import Fittable1DModel, Parameter
class Gaussian1D(Fittable1DModel):
n_inputs = 1
n_outputs = 1
amplitude = Parameter()
mean = Parameter()
stddev = Parameter()
这个 n_inputs 和 n_outputs 类属性必须是整数,指示为评估模型而输入的独立变量的数量及其返回的输出数量。输入和输出的标签, inputs 和 outputs ,将自动生成。可以通过在类中指定所需的值来覆盖默认值 __init__ 方法,调用后 super . outputs 和 inputs 必须是长度为的字符串元组 n_outputs 和 n_inputs 分别。输出可能与输入具有相同的标签(例如。 inputs = ('x', 'y') 和 outputs = ('x', 'y') ). 但是,输入不能相互冲突(例如。 inputs = ('x', 'x') 不正确),输出也是如此。
建模包中有两个有用的基类,可以用来避免指定 n_inputs 和 n_outputs 最常见的型号。这些是 Fittable1DModel 和 Fittable2DModel . 例如,实际 Gaussian1D 模型是 Fittable1DModel . 这有助于通过不必指定 n_inputs , n_outputs , inputs 和 outputs 对于许多模型(例如,可以通过Gaussian1D的链接查看其源代码)。
拟合模型可以是线性的,也可以是回归意义上的非线性模型。的默认值 linear 属性是 False . 线性模型应定义 linear 将属性分类为 True . 因为这个模型是非线性的,我们可以使用默认值。
继承自 Fittable1DModel 有 Model._separable 已设置为属性 True . 所有其他模型都应定义此属性以指示 模型可分性 .
接下来,提供调用 evaluate 评估模型和 fit_deriv ,以计算其相对于参数的导数。这些可能是正常的方法, classmethod 或 staticmethod ,尽管惯例是使用 staticmethod 当函数不依赖于对象的任何其他属性时(即,它不引用 self )或者类的任何其他属性 classmethod . 求值方法将所有输入坐标作为单独的参数,并且所有模型参数的顺序与它们将被列出的顺序相同 param_names .
对于这个例子:
@staticmethod
def evaluate(x, amplitude, mean, stddev):
return amplitude * np.exp((-(1 / (2. * stddev**2)) * (x - mean)**2))
应该明确指出 evaluate 方法必须设计为将模型的参数值作为参数。这似乎与参数值已经通过模型的属性(例如。 model.amplitude ). 但是,将参数值直接传递给 evaluate 是一种在许多情况下使用它的更有效的方法,例如安装。
模型的用户通常不会使用 evaluate 直接。相反,他们创建模型的一个实例,并在某些输入上调用它。这个 __call__ 模型使用方法 evaluate 在内部,但用户不需要知道。违约 __call__ 实现还处理一些细节,比如在尝试评估模型之前检查输入是否正确格式化并遵循Numpy的广播规则。
喜欢 evaluate , the fit_deriv 方法将所有坐标和所有参数值作为参数作为输入。可以选择计算非线性模型的数值导数,在这种情况下 fit_deriv 方法应该是 None ::
@staticmethod
def fit_deriv(x, amplitude, mean, stddev):
d_amplitude = np.exp(- 0.5 / stddev**2 * (x - mean)**2)
d_mean = (amplitude *
np.exp(- 0.5 / stddev**2 * (x - mean)**2) *
(x - mean) / stddev**2)
d_stddev = (2 * amplitude *
np.exp(- 0.5 / stddev**2 * (x - mean)**2) *
(x - mean)**2 / stddev**3)
return [d_amplitude, d_mean, d_stddev]
请注意,我们做到了 not 必须定义 __init__ 方法或 __call__ 我们模型的方法。对于大多数型号 __init__ 遵循相同的模式,将参数值作为位置参数,后跟几个可选的关键字参数(约束等)。建模框架自动生成 __init__ 对于具有正确调用签名的类(通过调用为您自己查看 help(Gaussian1D.__init__) 在我们刚刚定义的示例模型上)。
在某些情况下,可能需要定义一个自定义项 __init__ . 例如, Gaussian2D model takes an optional cov_matrix argument which can be used as an alternative way to specify the x/y_stddev and theta parameters. This is perfectly valid so long as the _ _init\uu``为实际参数确定适当的值,然后调用super ``__init__ 用标准参数。示意图如下:
def __init__(self, amplitude, x_mean, y_mean, x_stddev=None,
y_stddev=None, theta=None, cov_matrix=None, **kwargs):
# The **kwargs here should be understood as other keyword arguments
# accepted by the basic Model.__init__ (such as constraints)
if cov_matrix is not None:
# Set x/y_stddev and theta from the covariance matrix
x_stddev = ...
y_stddev = ...
theta = ...
# Don't pass on cov_matrix since it doesn't mean anything to the base
# class
super().__init__(amplitude, x_mean, y_mean, x_stddev, y_stddev, theta,
**kwargs)
完整例子#
import numpy as np
from astropy.modeling import Fittable1DModel, Parameter
class Gaussian1D(Fittable1DModel):
amplitude = Parameter()
mean = Parameter()
stddev = Parameter()
@staticmethod
def evaluate(x, amplitude, mean, stddev):
return amplitude * np.exp((-(1 / (2. * stddev**2)) * (x - mean)**2))
@staticmethod
def fit_deriv(x, amplitude, mean, stddev):
d_amplitude = np.exp((-(1 / (stddev**2)) * (x - mean)**2))
d_mean = (2 * amplitude *
np.exp((-(1 / (stddev**2)) * (x - mean)**2)) *
(x - mean) / (stddev**2))
d_stddev = (2 * amplitude *
np.exp((-(1 / (stddev**2)) * (x - mean)**2)) *
((x - mean)**2) / (stddev**3))
return [d_amplitude, d_mean, d_stddev]
线型模型的完整示例#
此示例演示了模型类的另一个可选功能,即 逆 . 安 inverse 实施应该是 property 它返回一个新的模型实例(不一定与被反转的模型属于同一个类),该实例计算该模型的逆,因此对于某些具有逆的模型实例, model.inverse(model(*input)) == input .
import numpy as np
from astropy.modeling import Fittable1DModel, Parameter
class LineModel(Fittable1DModel):
slope = Parameter()
intercept = Parameter()
linear = True
@staticmethod
def evaluate(x, slope, intercept):
return slope * x + intercept
@staticmethod
def fit_deriv(x, slope, intercept):
d_slope = x
d_intercept = np.ones_like(x)
return [d_slope, d_intercept]
@property
def inverse(self):
new_slope = self.slope ** -1
new_intercept = -self.intercept / self.slope
return LineModel(slope=new_slope, intercept=new_intercept)
备注
上面的示例基本上等同于内置的 Linear1D 模型。

