卷积和滤波 (astropy.convolution )#
介绍#
astropy.convolution 提供卷积函数和内核,与SciPy相比有改进 scipy.ndimage 卷积程序,包括:
正确处理NaN值(在卷积期间忽略它们,并用插值值替换NaN像素)
直接和快速傅里叶变换(FFT)版本
天文学中常用的内置内核
下面的缩略图显示了在包含NaN值的天文图像上SciPy和Astropy卷积函数之间的差异。对于任何NaN值的内核大小区域内的所有像素,Scipy的函数都会返回NaN,这通常不是预期的结果。
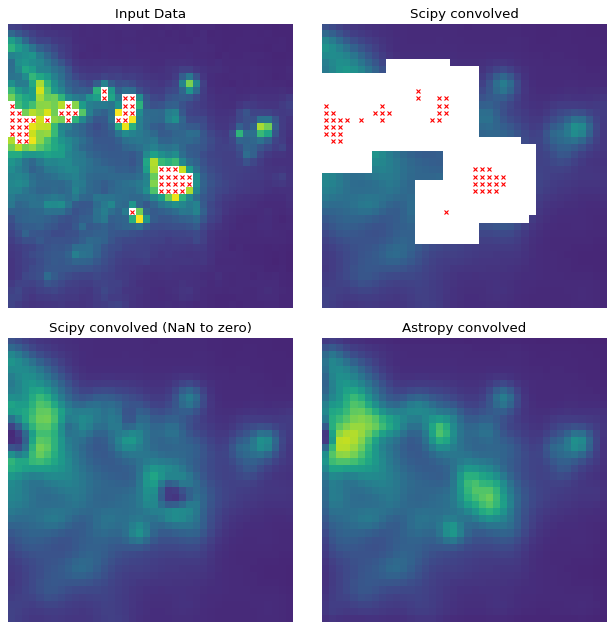
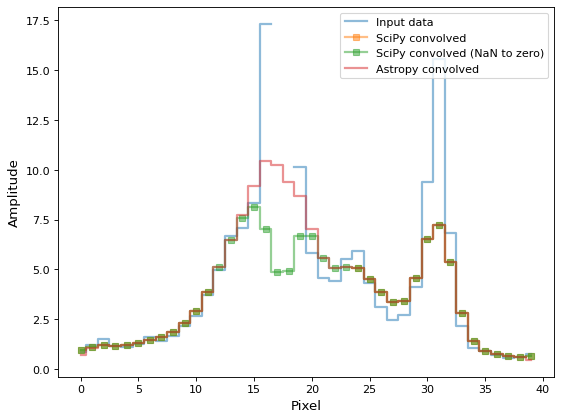
入门#
提供两个卷积函数。它们被导入为:
from astropy.convolution import convolve, convolve_fft
它们都用作:
result = convolve(image, kernel)
result = convolve_fft(image, kernel)
convolve() 实现为直接卷积算法,而 convolve_fft() 使用快速傅立叶变换(FFT)。因此,前者更适合于小内核,而后者对于较大的内核更有效。
例子#
要使用用户指定的内核对1D数据集进行卷积,可以执行以下操作:
>>> from astropy.convolution import convolve
>>> convolve([1, 4, 5, 6, 5, 7, 8], [0.2, 0.6, 0.2])
array([1.4, 3.6, 5. , 5.6, 5.6, 6.8, 6.2])
这个 boundary 关键字确定如何将输入数组扩展到其边界之外。缺省值为 'fill' ,表示数组边界外的值被设置为输入 fill_value (默认为0.0)。设置 boundary='extend' 使用常量外推将边缘附近的值扩展到边界之外。最后的值是假定第一个点以下的任何值都是 1 ,任何高于最后一个点的值都是 8 **
>>> from astropy.convolution import convolve
>>> convolve([1, 4, 5, 6, 5, 7, 8], [0.2, 0.6, 0.2], boundary='extend')
array([1.6, 3.6, 5. , 5.6, 5.6, 6.8, 7.8])
有关边界处理的更详细讨论,请参见 使用卷积函数 。
例子#
卷积模块还包括可以导入的内置内核,例如:
>>> from astropy.convolution import Gaussian1DKernel
要使用内核,首先创建一个特定的内核实例:
>>> gauss = Gaussian1DKernel(stddev=2)
gauss 不是数组,而是内核对象。可以使用以下命令检索基础数组:
>>> gauss.array
array([6.69162896e-05, 4.36349021e-04, 2.21596317e-03, 8.76430436e-03,
2.69959580e-02, 6.47599366e-02, 1.20987490e-01, 1.76035759e-01,
1.99474648e-01, 1.76035759e-01, 1.20987490e-01, 6.47599366e-02,
2.69959580e-02, 8.76430436e-03, 2.21596317e-03, 4.36349021e-04,
6.69162896e-05])
然后可以在调用时直接使用内核 convolve() :
(Source code, png, svg, pdf)
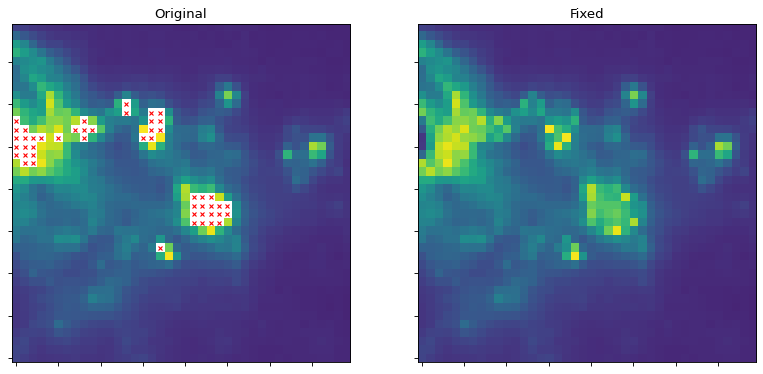
使用 astropy 的卷积来替换坏数据#
astropy 的卷积方法可以用来用从相邻数据中插值的值替换坏数据。基于核的插值对于处理含有少量坏像素的图像或对稀疏采样的图像进行插值非常有用。
插值工具的实现和用途如下:
from astropy.convolution import interpolate_replace_nans
result = interpolate_replace_nans(image, kernel)
您可能希望使用基于内核的插值的一些上下文包括:
像素饱和的图像。一般来说,这些是成像区域中强度最高的区域,插值值不可靠,但这对于显示目的是有用的。
带有标记像素的图像(例如,受宇宙射线或其他伪信号影响的一些小区域需要标记这些像素)。如果受影响的区域足够小,所得到的插值将对源统计数据产生较小的影响,并允许在结果数据上运行健壮的源查找算法。
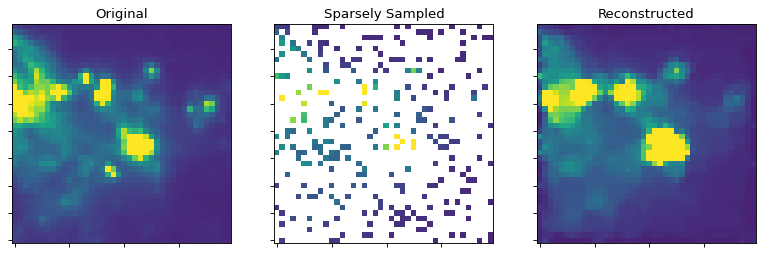
稀疏采样的图像,例如用单像素探测器构建的图像。这样的图像只有几个离散点在整个成像区域内采样,但是仍然可以构造出扩展天空发射的近似值。
备注
必须注意确保内核足够大,足以完全覆盖NaN值的潜在相邻区域。安 AstropyUserWarning 如果在后卷积检测到NaN值,则引发,在这种情况下,应增大内核大小。
例子#
下面的脚本显示了填充标记的像素的内核插值示例:
(Source code, png, svg, pdf)
例子#
这个脚本展示了这种技术从稀疏采样重建图像的能力。请注意,图像并不完美:点状源有时会丢失,但扩展的结构可以通过眼睛很好地恢复。
(Source code, png, svg, pdf)
使用 astropy.convolution#
性能提示#
这个 convolve() 函数最适合于较小的内核,但对于较大的内核可能会变得非常慢。在这种情况下,可以考虑使用 convolve_fft() (不过请注意,此函数使用更多内存,并考虑不同的填充选项)。
