提升性能#
在本教程的这一部分中,我们将研究如何加快在Pandas上运行某些函数的速度 DataFrame 使用三种不同的技术:Cython、Numba和 pandas.eval() 。当我们在一个测试函数上使用Cython和Numba时,我们将看到速度提高了约200。 DataFrame 。使用 pandas.eval() 我们将把求和速度提高~2个数量级。
备注
除了遵循本教程中的步骤外,强烈建议对增强性能感兴趣的用户安装 recommended dependencies 对于Pandas来说。这些依赖项通常在默认情况下不会安装,但如果存在,则会提高速度。
Cython(为Pandas编写C扩展)#
对于许多用例,用纯Python和NumPy编写Pandas就足够了。然而,在一些计算量很大的应用程序中,可以通过将工作分流到 cython 。
本教程假定您已经在Python语言中进行了尽可能多的重构,例如,通过尝试删除for循环和使用NumPy矢量化。它总是值得首先在Python中进行优化。
本教程将介绍一个“典型”的过程,即对速度较慢的计算进行Cython化。我们使用一种 example from the Cython documentation 但在Pandas的背景下。我们最终的Cython化解决方案比纯Python解决方案快100倍左右。
纯粹的 Python#
我们有一个 DataFrame 我们想要按行对其应用函数。
In [1]: df = pd.DataFrame(
...: {
...: "a": np.random.randn(1000),
...: "b": np.random.randn(1000),
...: "N": np.random.randint(100, 1000, (1000)),
...: "x": "x",
...: }
...: )
...:
In [2]: df
Out[2]:
a b N x
0 0.469112 -0.218470 585 x
1 -0.282863 -0.061645 841 x
2 -1.509059 -0.723780 251 x
3 -1.135632 0.551225 972 x
4 1.212112 -0.497767 181 x
.. ... ... ... ..
995 -1.512743 0.874737 374 x
996 0.933753 1.120790 246 x
997 -0.308013 0.198768 157 x
998 -0.079915 1.757555 977 x
999 -1.010589 -1.115680 770 x
[1000 rows x 4 columns]
下面是纯Python中的函数:
In [3]: def f(x):
...: return x * (x - 1)
...:
In [4]: def integrate_f(a, b, N):
...: s = 0
...: dx = (b - a) / N
...: for i in range(N):
...: s += f(a + i * dx)
...: return s * dx
...:
我们通过使用以下工具来实现我们的结果 DataFrame.apply() (按行):
In [5]: %timeit df.apply(lambda x: integrate_f(x["a"], x["b"], x["N"]), axis=1)
69.5 ms +- 141 us per loop (mean +- std. dev. of 7 runs, 10 loops each)
但显然,这对我们来说还不够快。让我们查看一下,看看在此操作期间(限于最耗时的四个调用)时间都花在了哪里 prun ipython magic function :
In [6]: %prun -l 4 df.apply(lambda x: integrate_f(x["a"], x["b"], x["N"]), axis=1) # noqa E999
621395 function calls (621371 primitive calls) in 0.153 seconds
Ordered by: internal time
List reduced from 221 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
1000 0.087 0.000 0.131 0.000 <ipython-input-4-c2a74e076cf0>:1(integrate_f)
552423 0.044 0.000 0.044 0.000 <ipython-input-3-c138bdd570e3>:1(f)
3000 0.004 0.000 0.016 0.000 series.py:952(__getitem__)
3000 0.002 0.000 0.008 0.000 series.py:1058(_get_value)
到目前为止,大部分时间都是在室内度过的 integrate_f 或 f 因此,我们将集中精力将这两个功能系统化。
素色Cython#
首先,我们需要将Cython魔术函数导入到IPython中:
In [7]: %load_ext Cython
现在,让我们简单地将我们的函数按原样复制到Cython中(这里的后缀用于区分函数版本):
In [8]: %%cython
...: def f_plain(x):
...: return x * (x - 1)
...: def integrate_f_plain(a, b, N):
...: s = 0
...: dx = (b - a) / N
...: for i in range(N):
...: s += f_plain(a + i * dx)
...: return s * dx
...:
备注
如果你在将上面的内容粘贴到你的IPython时遇到了麻烦,你可能需要使用尖端的IPython来粘贴,才能很好地玩细胞魔术。
In [9]: %timeit df.apply(lambda x: integrate_f_plain(x["a"], x["b"], x["N"]), axis=1)
37.6 ms +- 48.8 us per loop (mean +- std. dev. of 7 runs, 10 loops each)
这已经削减了三分之一,对于简单的复制和粘贴来说还不算太差。
添加类型#
我们只需提供类型信息即可获得另一个巨大的改进:
In [10]: %%cython
....: cdef double f_typed(double x) except? -2:
....: return x * (x - 1)
....: cpdef double integrate_f_typed(double a, double b, int N):
....: cdef int i
....: cdef double s, dx
....: s = 0
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....:
In [11]: %timeit df.apply(lambda x: integrate_f_typed(x["a"], x["b"], x["N"]), axis=1)
7.6 ms +- 13.9 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
现在,我们在说话了!它现在比最初的Python实现快了十多倍,而我们没有 真的 修改了代码。让我们再来看看是什么在消耗时间:
In [12]: %prun -l 4 df.apply(lambda x: integrate_f_typed(x["a"], x["b"], x["N"]), axis=1)
68972 function calls (68948 primitive calls) in 0.022 seconds
Ordered by: internal time
List reduced from 220 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
3000 0.004 0.000 0.016 0.000 series.py:952(__getitem__)
3000 0.002 0.000 0.008 0.000 series.py:1058(_get_value)
3000 0.002 0.000 0.003 0.000 base.py:3735(get_loc)
3000 0.002 0.000 0.002 0.000 indexing.py:2572(check_deprecated_indexers)
使用ndarray#
这部电视剧叫得太多了!它正在创造一种 Series 并从索引和序列调用GET(每行调用三次)。在Python中,函数调用的开销很大,所以也许我们可以通过将Apply部分变为Cython化来最小化这些开销。
备注
我们现在将ndarray传递给Cython函数,幸运的是,Cython可以很好地处理NumPy。
In [13]: %%cython
....: cimport numpy as np
....: import numpy as np
....: cdef double f_typed(double x) except? -2:
....: return x * (x - 1)
....: cpdef double integrate_f_typed(double a, double b, int N):
....: cdef int i
....: cdef double s, dx
....: s = 0
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....: cpdef np.ndarray[double] apply_integrate_f(np.ndarray col_a, np.ndarray col_b,
....: np.ndarray col_N):
....: assert (col_a.dtype == np.float_
....: and col_b.dtype == np.float_ and col_N.dtype == np.int_)
....: cdef Py_ssize_t i, n = len(col_N)
....: assert (len(col_a) == len(col_b) == n)
....: cdef np.ndarray[double] res = np.empty(n)
....: for i in range(len(col_a)):
....: res[i] = integrate_f_typed(col_a[i], col_b[i], col_N[i])
....: return res
....:
实现很简单,它在各行上创建一个由零和循环组成的数组,应用我们的 integrate_f_typed ,并将其放入零数组中。
警告
你可以的 未通过 一个 Series 直接作为 ndarray Cython函数的类型化参数。而是传递实际的 ndarray 使用 Series.to_numpy() 。原因是Cython定义是特定于ndarray的,而不是传递的 Series 。
因此,请不要这样做:
apply_integrate_f(df["a"], df["b"], df["N"])
而是使用 Series.to_numpy() 要获得潜在的 ndarray :
apply_integrate_f(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
备注
像这样的循环将是 极 在Python中速度很慢,但在Cython中对NumPy数组进行循环是 fast 。
In [14]: %timeit apply_integrate_f(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
754 us +- 514 ns per loop (mean +- std. dev. of 7 runs, 1,000 loops each)
我们又有了很大的进步。让我们再检查一下时间都花在哪里了:
In [15]: %prun -l 4 apply_integrate_f(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
85 function calls in 0.001 seconds
Ordered by: internal time
List reduced from 24 to 4 due to restriction <4>
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.001 0.001 0.001 0.001 {built-in method _cython_magic_a1d6ab09d3160a3feb7d9dd78f5a52d6.apply_integrate_f}
1 0.000 0.000 0.001 0.001 {built-in method builtins.exec}
3 0.000 0.000 0.000 0.000 frame.py:3488(__getitem__)
3 0.000 0.000 0.000 0.000 base.py:432(to_numpy)
正如人们所料,现在大部分时间都花在 apply_integrate_f 因此,如果我们想要提高效率,我们必须继续在这里集中我们的努力。
更先进的技术#
仍有改善的希望。以下是使用一些更高级的Cython技术的示例:
In [16]: %%cython
....: cimport cython
....: cimport numpy as np
....: import numpy as np
....: cdef np.float64_t f_typed(np.float64_t x) except? -2:
....: return x * (x - 1)
....: cpdef np.float64_t integrate_f_typed(np.float64_t a, np.float64_t b, np.int64_t N):
....: cdef np.int64_t i
....: cdef np.float64_t s = 0.0, dx
....: dx = (b - a) / N
....: for i in range(N):
....: s += f_typed(a + i * dx)
....: return s * dx
....: @cython.boundscheck(False)
....: @cython.wraparound(False)
....: cpdef np.ndarray[np.float64_t] apply_integrate_f_wrap(
....: np.ndarray[np.float64_t] col_a,
....: np.ndarray[np.float64_t] col_b,
....: np.ndarray[np.int64_t] col_N
....: ):
....: cdef np.int64_t i, n = len(col_N)
....: assert len(col_a) == len(col_b) == n
....: cdef np.ndarray[np.float64_t] res = np.empty(n, dtype=np.float64)
....: for i in range(n):
....: res[i] = integrate_f_typed(col_a[i], col_b[i], col_N[i])
....: return res
....:
In [17]: %timeit apply_integrate_f_wrap(df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy())
659 us +- 2.68 us per loop (mean +- std. dev. of 7 runs, 1,000 loops each)
速度更快,但需要注意的是,我们的Cython代码中的错误(例如,Off-by-one错误)可能会导致段错误,因为没有检查内存访问。有关详细信息,请参阅 boundscheck and wraparound, see the Cython docs on compiler directives 。
Numba(JIT编译)#
静态编译Cython代码的另一种方法是使用动态实时(JIT)编译器 Numba 。
Numba允许您编写纯Python函数,该函数可以JIT编译为本机机器指令,其性能类似于C、C++和Fortran,方法是用 @jit 。
Numba的工作方式是在导入时、运行时或静态(使用附带的pycc工具)使用LLVM编译器基础设施生成优化的机器码。Numba支持编译在CPU或GPU硬件上运行的Python,并设计为与Python科学软件堆栈集成。
备注
这个 @jit compilation will add overhead to the runtime of the function, so performance benefits may not be realized especially when using small data sets. Consider caching 函数,以避免每次运行函数时的编译开销。
Numba在Pandas身上有两种用途:
指定
engine="numba"选择Pandas方法中的关键字定义您自己的用来装饰的Python函数
@jit并将基础NumPy数组Series或DataFrame(使用to_numpy())添加到函数中
PandasNumba引擎#
如果安装了Numba,则可以指定 engine="numba" 在选择Pandas方法时,使用Numba执行该方法。支持以下内容的方法 engine="numba" 还将拥有一个 engine_kwargs 关键字,该关键字接受允许用户指定 "nogil" , "nopython" 和 "parallel" 包含布尔值的键以传递到 @jit 装饰师。如果 engine_kwargs 未指定,则默认为 {{"nogil": False, "nopython": True, "parallel": False}} 除非另有说明。
在性能方面, 第一次使用Numba引擎运行函数会很慢 因为Numba会有一些函数编译开销。但是,JIT编译后的函数会被缓存,因此后续调用会很快。一般而言,Numba引擎具有较大的数据点数量(例如,100+百万)。
In [1]: data = pd.Series(range(1_000_000)) # noqa: E225
In [2]: roll = data.rolling(10)
In [3]: def f(x):
...: return np.sum(x) + 5
# Run the first time, compilation time will affect performance
In [4]: %timeit -r 1 -n 1 roll.apply(f, engine='numba', raw=True)
1.23 s ± 0 ns per loop (mean ± std. dev. of 1 run, 1 loop each)
# Function is cached and performance will improve
In [5]: %timeit roll.apply(f, engine='numba', raw=True)
188 ms ± 1.93 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [6]: %timeit roll.apply(f, engine='cython', raw=True)
3.92 s ± 59 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
如果您的计算硬件包含多个CPU,则可以通过设置 parallel 至 True 利用1个以上的CPU。在内部,Pandas利用Numba来并行计算 DataFrame ;因此,此性能优势仅对 DataFrame 有大量的柱子。
In [1]: import numba
In [2]: numba.set_num_threads(1)
In [3]: df = pd.DataFrame(np.random.randn(10_000, 100))
In [4]: roll = df.rolling(100)
In [5]: %timeit roll.mean(engine="numba", engine_kwargs={"parallel": True})
347 ms ± 26 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [6]: numba.set_num_threads(2)
In [7]: %timeit roll.mean(engine="numba", engine_kwargs={"parallel": True})
201 ms ± 2.97 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
自定义函数示例#
用以下元素修饰的自定义Python函数 @jit 通过将Pandas对象的NumPy数组表示形式传递给 to_numpy() 。
import numba
@numba.jit
def f_plain(x):
return x * (x - 1)
@numba.jit
def integrate_f_numba(a, b, N):
s = 0
dx = (b - a) / N
for i in range(N):
s += f_plain(a + i * dx)
return s * dx
@numba.jit
def apply_integrate_f_numba(col_a, col_b, col_N):
n = len(col_N)
result = np.empty(n, dtype="float64")
assert len(col_a) == len(col_b) == n
for i in range(n):
result[i] = integrate_f_numba(col_a[i], col_b[i], col_N[i])
return result
def compute_numba(df):
result = apply_integrate_f_numba(
df["a"].to_numpy(), df["b"].to_numpy(), df["N"].to_numpy()
)
return pd.Series(result, index=df.index, name="result")
In [4]: %timeit compute_numba(df)
1000 loops, best of 3: 798 us per loop
在本例中,使用Numba比使用Cython更快。
Numba还可以用于编写矢量化函数,这些函数不需要用户显式循环遍历向量的观测值;矢量化函数将自动应用于每行。考虑以下将每个观测值加倍的示例:
import numba
def double_every_value_nonumba(x):
return x * 2
@numba.vectorize
def double_every_value_withnumba(x): # noqa E501
return x * 2
# Custom function without numba
In [5]: %timeit df["col1_doubled"] = df["a"].apply(double_every_value_nonumba) # noqa E501
1000 loops, best of 3: 797 us per loop
# Standard implementation (faster than a custom function)
In [6]: %timeit df["col1_doubled"] = df["a"] * 2
1000 loops, best of 3: 233 us per loop
# Custom function with numba
In [7]: %timeit df["col1_doubled"] = double_every_value_withnumba(df["a"].to_numpy())
1000 loops, best of 3: 145 us per loop
注意事项#
Numba最擅长加速将数值函数应用于NumPy数组的函数。如果你试图 @jit a function that contains unsupported Python 或 NumPy 代码,编译将恢复 object mode 这很可能不会加快您的功能。如果您希望Numba在无法以加快代码速度的方式编译函数时抛出错误,请将参数传递给Numba nopython=True (e.g. @jit(nopython=True)). For more on troubleshooting Numba modes, see the Numba troubleshooting page 。
使用 parallel=True (e.g. @jit(parallel=True)) may result in a SIGABRT if the threading layer leads to unsafe behavior. You can first specify a safe threading layer 在使用运行JIT函数之前 parallel=True 。
通常,如果您遇到段错误 (SIGSEGV )在使用Numba时,请将问题报告给 Numba issue tracker.
表达式求值方式 eval()#
顶层函数 pandas.eval() 实现表达式求值 Series 和 DataFrame 对象。
备注
从使用中受益 eval() 您需要安装 numexpr 。请参阅 recommended dependencies section 了解更多详细信息。
使用的要点是 eval() 对于表达式求值,而不是普通的,Python有两个方面:1)大 DataFrame 对象的求值效率更高,2)底层引擎一次计算大型算术和布尔表达式(默认情况下 numexpr 用于评估)。
备注
您不应该使用 eval() 用于简单表达式或涉及小DataFrame的表达式。事实上, eval() 对于较小的表达式/对象,速度比普通的ol‘Python慢许多个数量级。一个好的经验法则是只使用 eval() 当你有一个 DataFrame 有10,000多行。
eval() 支持引擎支持的所有算术表达式,以及一些仅在PANDA中可用的扩展。
备注
框架越大,表达式越大,使用 eval() 。
支持的语法#
这些操作由支持 pandas.eval() :
除左移以外的算术运算 (
<<)和右移 (>>)运算符,例如,df + 2 * pi / s ** 4 % 42 - the_golden_ratio比较操作,包括链式比较,例如,
2 < df < df2布尔运算,例如,
df < df2 and df3 < df4 or not df_boollistandtupleliterals, e.g.,[1, 2]or(1, 2)属性访问,例如,
df.a下标表达式,例如,
df[0]简单的变量评估,例如,
pd.eval("df")(这不是很有用)数学函数:
sin,cos,exp,log,expm1,log1p,sqrt,sinh,cosh,tanh,arcsin,arccos,arctan,arccosh,arcsinh,arctanh,abs,arctan2和log10。
此Python语法为 not 允许:
表达式
数学函数以外的函数调用。
is/is not运营if表达式lambda表达式list/set/``口述``理解字面意思
dict和set表达式yield表达式生成器表达式
仅包含标量值的布尔表达式
报表
eval() 示例#
pandas.eval() 可以很好地处理包含大型数组的表达式。
首先,让我们创建几个大小合适的数组来处理:
In [18]: nrows, ncols = 20000, 100
In [19]: df1, df2, df3, df4 = [pd.DataFrame(np.random.randn(nrows, ncols)) for _ in range(4)]
现在,让我们比较一下使用简单的Python将它们加在一起与使用它们 eval() :
In [20]: %timeit df1 + df2 + df3 + df4
10.9 ms +- 201 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [21]: %timeit pd.eval("df1 + df2 + df3 + df4")
10.9 ms +- 71.2 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
现在让我们做同样的事情,但进行比较:
In [22]: %timeit (df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)
6.36 ms +- 34.1 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [23]: %timeit pd.eval("(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)")
7.15 ms +- 62.2 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
eval() 也适用于未对齐的Pandas对象:
In [24]: s = pd.Series(np.random.randn(50))
In [25]: %timeit df1 + df2 + df3 + df4 + s
17.5 ms +- 197 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [26]: %timeit pd.eval("df1 + df2 + df3 + df4 + s")
18.4 ms +- 87.8 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
备注
操作,如
1 and 2 # would parse to 1 & 2, but should evaluate to 2 3 or 4 # would parse to 3 | 4, but should evaluate to 3 ~1 # this is okay, but slower when using eval
应该在Python语言中执行。如果尝试对非类型的标量操作数执行任何布尔/按位运算,将引发异常 bool 或 np.bool_ 。同样,您应该在普通的Python中执行这些类型的操作。
这个 DataFrame.eval() 方法#
除了最高层 pandas.eval() 函数,您还可以在 DataFrame 。
In [27]: df = pd.DataFrame(np.random.randn(5, 2), columns=["a", "b"])
In [28]: df.eval("a + b")
Out[28]:
0 -0.246747
1 0.867786
2 -1.626063
3 -1.134978
4 -1.027798
dtype: float64
任何有效的表达式 pandas.eval() 表达式也是有效的 DataFrame.eval() 表达式的附加好处是,您不必在 DataFrame 添加到您感兴趣评估的列。
此外,您还可以在表达式中执行列赋值。这允许 公式化评价 。赋值目标可以是新的列名,也可以是现有的列名,并且必须是有效的Python标识符。
这个 inplace 关键字确定此赋值是否将在原始 DataFrame 或者返回一份包含新专栏的副本。
In [29]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [30]: df.eval("c = a + b", inplace=True)
In [31]: df.eval("d = a + b + c", inplace=True)
In [32]: df.eval("a = 1", inplace=True)
In [33]: df
Out[33]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
什么时候 inplace 设置为 False ,默认情况下,是 DataFrame 返回新的或修改过的列,原始框架保持不变。
In [34]: df
Out[34]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
In [35]: df.eval("e = a - c", inplace=False)
Out[35]:
a b c d e
0 1 5 5 10 -4
1 1 6 7 14 -6
2 1 7 9 18 -8
3 1 8 11 22 -10
4 1 9 13 26 -12
In [36]: df
Out[36]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
为了方便起见,可以使用多行字符串执行多个赋值。
In [37]: df.eval(
....: """
....: c = a + b
....: d = a + b + c
....: a = 1""",
....: inplace=False,
....: )
....:
Out[37]:
a b c d
0 1 5 6 12
1 1 6 7 14
2 1 7 8 16
3 1 8 9 18
4 1 9 10 20
在标准的Python中,等效项将是
In [38]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [39]: df["c"] = df["a"] + df["b"]
In [40]: df["d"] = df["a"] + df["b"] + df["c"]
In [41]: df["a"] = 1
In [42]: df
Out[42]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
这个 DataFrame.query 方法具有一个 inplace 关键字,该关键字确定查询是否修改原始框架。
In [43]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [44]: df.query("a > 2")
Out[44]:
a b
3 3 8
4 4 9
In [45]: df.query("a > 2", inplace=True)
In [46]: df
Out[46]:
a b
3 3 8
4 4 9
局部变量#
你必须 显式引用 要在表达式中使用的任何局部变量,方法是将 @ 名称前面的字符。例如,
In [47]: df = pd.DataFrame(np.random.randn(5, 2), columns=list("ab"))
In [48]: newcol = np.random.randn(len(df))
In [49]: df.eval("b + @newcol")
Out[49]:
0 -0.173926
1 2.493083
2 -0.881831
3 -0.691045
4 1.334703
Name: b, dtype: float64
In [50]: df.query("b < @newcol")
Out[50]:
a b
0 0.863987 -0.115998
2 -2.621419 -1.297879
如果不在局部变量前面加上前缀 @ 时,Pandas会抛出一个异常,告诉您变量未定义。
在使用时 DataFrame.eval() 和 DataFrame.query() ,这允许您拥有一个局部变量和一个 DataFrame 表达式中具有相同名称的列。
In [51]: a = np.random.randn()
In [52]: df.query("@a < a")
Out[52]:
a b
0 0.863987 -0.115998
In [53]: df.loc[a < df["a"]] # same as the previous expression
Out[53]:
a b
0 0.863987 -0.115998
使用 pandas.eval() 您不能使用 @ 前缀 完全没有 ,因为它不是在那个上下文中定义的。如果你尝试使用Pandas会让你知道这一点 @ 在顶级调用中 pandas.eval() 。例如,
In [54]: a, b = 1, 2
In [55]: pd.eval("@a + b")
Traceback (most recent call last):
File /usr/local/lib/python3.10/dist-packages/IPython/core/interactiveshell.py:3397 in run_code
exec(code_obj, self.user_global_ns, self.user_ns)
Input In [55] in <cell line: 1>
pd.eval("@a + b")
File /usr/local/lib/python3.10/dist-packages/pandas-1.5.0.dev0+697.gf9762d8f52-py3.10-linux-x86_64.egg/pandas/core/computation/eval.py:339 in eval
_check_for_locals(expr, level, parser)
File /usr/local/lib/python3.10/dist-packages/pandas-1.5.0.dev0+697.gf9762d8f52-py3.10-linux-x86_64.egg/pandas/core/computation/eval.py:163 in _check_for_locals
raise SyntaxError(msg)
File <string>
SyntaxError: The '@' prefix is not allowed in top-level eval calls.
please refer to your variables by name without the '@' prefix.
在这种情况下,您应该简单地引用变量,就像在标准的Python中一样。
In [56]: pd.eval("a + b")
Out[56]: 3
pandas.eval() 解析器#
您可以使用两种不同的解析器和两种不同的引擎作为后端。
默认设置 'pandas' Parser允许更直观的语法来表达类似查询的操作(比较、合取和析取)。尤其是, & 和 | 运算符的优先级等于相应布尔运算的优先级 and 和 or 。
例如,上面的连词可以写成不带括号。或者,您可以使用 'python' 解析器,以强制执行严格的Python语义。
In [57]: expr = "(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)"
In [58]: x = pd.eval(expr, parser="python")
In [59]: expr_no_parens = "df1 > 0 & df2 > 0 & df3 > 0 & df4 > 0"
In [60]: y = pd.eval(expr_no_parens, parser="pandas")
In [61]: np.all(x == y)
Out[61]: True
同样的表达可以与单词“AND”连在一起 and 还有:
In [62]: expr = "(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)"
In [63]: x = pd.eval(expr, parser="python")
In [64]: expr_with_ands = "df1 > 0 and df2 > 0 and df3 > 0 and df4 > 0"
In [65]: y = pd.eval(expr_with_ands, parser="pandas")
In [66]: np.all(x == y)
Out[66]: True
这个 and 和 or 这里的运算符与在普通的Python中具有相同的优先级。
pandas.eval() 后端#
还有一个选项是让 eval() 操作方式与普通的老式 Python 完全相同。
备注
使用 'python' 发动机一般是 not 很有用,除了用它测试其他评估引擎之外。您将实现 no 性能优势使用 eval() 使用 engine='python' 而且实际上可能会招致性能上的打击。
您可以使用以下命令查看此内容 pandas.eval() 使用 'python' 引擎。它比在Python语言中计算相同的表达式要慢一些(不会太慢
In [67]: %timeit df1 + df2 + df3 + df4
10.8 ms +- 115 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
In [68]: %timeit pd.eval("df1 + df2 + df3 + df4", engine="python")
11.1 ms +- 42.8 us per loop (mean +- std. dev. of 7 runs, 100 loops each)
pandas.eval() 性能#
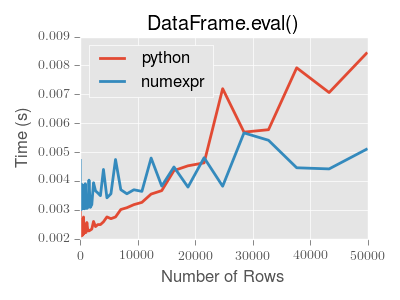
eval() is intended to speed up certain kinds of operations. In particular, those operations involving complex expressions with large DataFrame/Series 对象应该会看到显著的性能优势。这是一个显示运行时间的曲线图 pandas.eval() 作为计算中所涉及的帧大小的函数。这两条线路是两个不同的引擎。
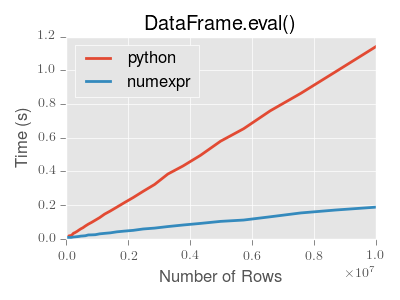
备注
对于较小对象(大约15k-20k行)的操作,使用普通的Python会更快:
此图是使用 DataFrame 包含3列,每列都包含使用 numpy.random.randn() 。
有关表达式求值的技术细节#
会导致对象数据类型或涉及DateTime操作的表达式(因为 NaT )必须在Python空间中求值。此行为的主要原因是为了保持与NumPy<1.7版本的向后兼容性。在这些版本的NumPy中,调用 ndarray.astype(str) 将截断长度超过60个字符的任何字符串。第二,我们不能通过 object 数组到 numexpr 因此,字符串比较必须在Python空间中进行计算。
结果是,这就是 only 适用于对象数据类型表达式。所以,如果你有一个表达式--例如
In [69]: df = pd.DataFrame(
....: {"strings": np.repeat(list("cba"), 3), "nums": np.repeat(range(3), 3)}
....: )
....:
In [70]: df
Out[70]:
strings nums
0 c 0
1 c 0
2 c 0
3 b 1
4 b 1
5 b 1
6 a 2
7 a 2
8 a 2
In [71]: df.query("strings == 'a' and nums == 1")
Out[71]:
Empty DataFrame
Columns: [strings, nums]
Index: []
比较的数字部分 (nums == 1 )将由以下机构评估 numexpr 。
总体而言, DataFrame.query()/pandas.eval() 将计算的子表达式 can 被评为 numexpr 以及必须在对用户透明的Python空间中求值的那些。这是通过从其参数和运算符推断表达式的结果类型来实现的。