统计数据 (scipy.stats )¶
引言¶
在本教程中,我们将讨论的许多(但当然不是全部)功能 scipy.stats 。这里的目的是向用户提供该软件包的工作知识。我们指的是 reference manual 了解更多详细信息。
注意:本文档正在编写中。
随机变量¶
已经实现了两个用于封装的通用分发类 continuous random variables 和 discrete random variables 。使用这些类已经实现了80多个连续随机变量(RV)和10个离散随机变量。除此之外,终端用户可以很容易地添加新的例程和分发。(如果您创建了一个,请贡献它。)
所有统计函数都位于子包中 scipy.stats 并且可以使用以下命令获得这些函数的相当完整的列表 info(stats) 。还可以从stats子程序包的文档字符串中获得可用的随机变量列表。
在下面的讨论中,我们主要关注连续房车。几乎所有东西都适用于离散变量,但我们在这里指出一些不同之处: 离散分布的特定点 。
在下面的代码示例中,我们假设 scipy.stats 包被导入为
>>> from scipy import stats
在某些情况下,我们假设单个对象被导入为
>>> from scipy.stats import norm
获取帮助¶
首先,所有发行版都附带帮助功能。为了仅获取一些基本信息,我们打印相关的文档字符串: print(stats.norm.__doc__) 。
要查找支持,即分布的上下限,请调用:
>>> print('bounds of distribution lower: %s, upper: %s' % norm.support())
bounds of distribution lower: -inf, upper: inf
我们可以使用以下命令列出发行版的所有方法和属性 dir(norm) 。事实证明,有些方法是私有的,尽管它们的名称不是这样命名的(它们的名称不以前导下划线开头),例如 veccdf ,仅可用于内部计算(这些方法在用户尝试使用它们时会发出警告,并将在某个时候被删除)。
要获取 real 主要方法,列举了冷冻配送的方法。(我们将解释 frozen 分布如下)。
>>> rv = norm()
>>> dir(rv) # reformatted
['__class__', '__delattr__', '__dict__', '__dir__', '__doc__', '__eq__',
'__format__', '__ge__', '__getattribute__', '__gt__', '__hash__',
'__init__', '__le__', '__lt__', '__module__', '__ne__', '__new__',
'__reduce__', '__reduce_ex__', '__repr__', '__setattr__', '__sizeof__',
'__str__', '__subclasshook__', '__weakref__', 'a', 'args', 'b', 'cdf',
'dist', 'entropy', 'expect', 'interval', 'isf', 'kwds', 'logcdf',
'logpdf', 'logpmf', 'logsf', 'mean', 'median', 'moment', 'pdf', 'pmf',
'ppf', 'random_state', 'rvs', 'sf', 'stats', 'std', 'var']
最后,我们可以通过自省获得可用的分发列表:
>>> dist_continu = [d for d in dir(stats) if
... isinstance(getattr(stats, d), stats.rv_continuous)]
>>> dist_discrete = [d for d in dir(stats) if
... isinstance(getattr(stats, d), stats.rv_discrete)]
>>> print('number of continuous distributions: %d' % len(dist_continu))
number of continuous distributions: 104
>>> print('number of discrete distributions: %d' % len(dist_discrete))
number of discrete distributions: 19
常用方法¶
连续房车的主要公共方法有:
房车:随机变量
PDF:概率密度函数
CDF:累积分布函数
SF:生存函数(1-CDF)
PPF:百分比点函数(CDF的倒数)
ISF:逆生存函数(SF的逆)
统计量:返回均值、方差、(Fisher‘s)偏斜或(Fisher’s)峰度
矩:分布的非中心矩
让我们以一辆普通的房车为例。
>>> norm.cdf(0)
0.5
要计算 cdf 在许多点上,我们可以传递一个列表或一个数字数组。
>>> norm.cdf([-1., 0, 1])
array([ 0.15865525, 0.5, 0.84134475])
>>> import numpy as np
>>> norm.cdf(np.array([-1., 0, 1]))
array([ 0.15865525, 0.5, 0.84134475])
因此,基本的方法,如 pdf , cdf ,等等,都是矢量化的。
还支持其他通常有用的方法:
>>> norm.mean(), norm.std(), norm.var()
(0.0, 1.0, 1.0)
>>> norm.stats(moments="mv")
(array(0.0), array(1.0))
要找出分布的中位数,我们可以使用百分比值函数 ppf ,它与 cdf :
>>> norm.ppf(0.5)
0.0
若要生成随机变量序列,请使用 size 关键字参数:
>>> norm.rvs(size=3)
array([-0.35687759, 1.34347647, -0.11710531]) # random
别这么想 norm.rvs(5) 生成5个变量:
>>> norm.rvs(5)
5.471435163732493 # random
这里, 5 由于没有关键字被解释为第一个可能的关键字参数, loc ,它是所有连续分布采用的一对关键字参数中的第一个。这就把我们带到了下一个小节的主题。
随机数生成¶
绘制随机数依赖于 numpy.random 包裹。在上面的示例中,特定的随机数流不能跨运行重现。要实现重现性,您可以显式 seed 一个随机数发生器。在NumPy中,生成器是 numpy.random.Generator 。下面是创建生成器的规范方法:
>>> from numpy.random import default_rng
>>> rng = default_rng()
固定种子可以这样做:
>>> # do NOT copy this value
>>> rng = default_rng(301439351238479871608357552876690613766)
警告
请勿使用此数字或公用值(如0)。仅使用一小组种子实例化较大的状态空间意味着有些初始状态是不可能达到的。如果每个人都使用这样的值,这会产生一些偏见。获取种子的一个好方法是使用 numpy.random.SeedSequence :
>>> from numpy.random import SeedSequence
>>> print(SeedSequence().entropy)
301439351238479871608357552876690613766 # random
这个 random_state 参数接受 numpy.random.Generator 类或一个整数,然后使用该整数为内部 Generator 对象:
>>> norm.rvs(size=5, random_state=rng)
array([ 0.47143516, -1.19097569, 1.43270697, -0.3126519 , -0.72058873]) # random
有关详细信息,请参阅 NumPy's documentation 。
要了解有关在SciPy中实现的随机数采样器的更多信息,请参阅 non-uniform random number sampling tutorial 和 quasi monte carlo tutorial
移位和缩放¶
所有连续分布都需要 loc 和 scale 作为调整分布的位置和尺度的关键字参数,例如,对于标准正态分布,位置是平均值,尺度是标准偏差。
>>> norm.stats(loc=3, scale=4, moments="mv")
(array(3.0), array(16.0))
在许多情况下,随机变量的标准化分布 X 是通过变换获得的 (X - loc) / scale 。默认值为 loc = 0 和 scale = 1 。
巧妙地使用 loc 和 scale 可以在许多方面帮助修改标准发行版。为了进一步说明缩放情况, cdf 均值为指数分布的房车 \(1/\lambda\) 由以下人员提供
通过应用上面的缩放规则,可以看到通过将 scale = 1./lambda 我们有合适的天平。
>>> from scipy.stats import expon
>>> expon.mean(scale=3.)
3.0
注解
采用形状参数的分布可能不只需要简单地应用 loc 和/或 scale 以达到所需的形式。例如,在给定恒定长度的向量的情况下,2-D向量长度的分布 \(R\) 被独立的N(0, \(\sigma^2\) )每种成分的偏差都是大米 (\(R/\sigma\) ,比例= \(\sigma\) )。第一个参数是需要随缩放的形状参数 \(x\) 。
均匀分布也很有趣:
>>> from scipy.stats import uniform
>>> uniform.cdf([0, 1, 2, 3, 4, 5], loc=1, scale=4)
array([ 0. , 0. , 0.25, 0.5 , 0.75, 1. ])
最后,从上一段中回想一下,我们剩下的问题是 norm.rvs(5) 。结果是,调用这样的分布时,第一个参数(即5)被传递以设置 loc 参数。让我们看看:
>>> np.mean(norm.rvs(5, size=500))
5.0098355106969992 # random
因此,要解释上一节示例的输出: norm.rvs(5) 生成具有平均值的单一正态分布随机变量 loc=5 ,因为默认设置 size=1 。
我们建议您设置 loc 和 scale 参数,方法是将值作为关键字而不是参数传递。在调用给定RV的多个方法时,可以使用 Freezing a Distribution ,如下所述。
形状参数¶
而一般的连续随机变量可以用 loc 和 scale 参数,某些分布需要附加的形状参数。例如,具有密度的伽马分布
需要Shape参数 \(a\) 。请注意该设置 \(\lambda\) 可以通过设置 scale 关键字至 \(1/\lambda\) 。
让我们检查伽马分布的形状参数的数量和名称。(从上面我们知道这应该是1。)
>>> from scipy.stats import gamma
>>> gamma.numargs
1
>>> gamma.shapes
'a'
现在,我们将形状变量的值设置为1来得到指数分布,这样我们就可以很容易地比较我们是否得到了预期的结果。
>>> gamma(1, scale=2.).stats(moments="mv")
(array(2.0), array(4.0))
请注意,我们还可以将形状参数指定为关键字:
>>> gamma(a=1, scale=2.).stats(moments="mv")
(array(2.0), array(4.0))
冻结分发¶
通过 loc 和 scale 一次又一次的关键词可能会变得相当麻烦。的概念 freezing 房车就是用来解决这些问题的。
>>> rv = gamma(1, scale=2.)
通过使用 rv 我们不再需要包括比例或形状参数。因此,分布的使用方式有两种,一种是将所有分布参数传递给每个方法调用(如前面所做的那样),另一种是冻结分布实例的参数。让我们检查一下:
>>> rv.mean(), rv.std()
(2.0, 2.0)
这确实是我们应该得到的。
广播¶
基本方法 pdf 等等,满足通常的麻木广播规则。例如,对于不同的概率和自由度,我们可以计算t分布上尾的临界值。
>>> stats.t.isf([0.1, 0.05, 0.01], [[10], [11]])
array([[ 1.37218364, 1.81246112, 2.76376946],
[ 1.36343032, 1.79588482, 2.71807918]])
这里,第一行包含10个自由度的临界值,第二行包含11个自由度(直径)的临界值。因此,广播规则给出的结果与调用 isf 两次:
>>> stats.t.isf([0.1, 0.05, 0.01], 10)
array([ 1.37218364, 1.81246112, 2.76376946])
>>> stats.t.isf([0.1, 0.05, 0.01], 11)
array([ 1.36343032, 1.79588482, 2.71807918])
如果具有概率的数组,即, [0.1, 0.05, 0.01] 以及自由度阵列,即, [10, 11, 12] ,具有相同的阵列形状,则使用逐元素匹配。例如,我们可以得到10d.f的10%的尾部,11d.f的5%的尾部。12d.f的1%的尾部。通过调用
>>> stats.t.isf([0.1, 0.05, 0.01], [10, 11, 12])
array([ 1.37218364, 1.79588482, 2.68099799])
离散分布的特定点¶
离散分布与连续分布的基本方法基本相同。然而, pdf 被概率质量函数取代 pmf ,没有可用的估计方法,如FIT,以及 scale 不是有效的关键字参数。位置参数、关键字 loc ,仍可用于移动分布。
CDF的计算需要额外注意。在连续分布的情况下,在大多数标准情况下,累积分布函数在(a,b)界内严格单调递增,因此具有唯一的逆。然而,离散分布的CDF是阶跃函数,因此逆CDF(即百分比点函数)需要不同的定义:
ppf(q) = min{x : cdf(x) >= q, x integer}
有关详细信息,请参阅文档 here 。
我们可以以超几何分布为例
>>> from scipy.stats import hypergeom
>>> [M, n, N] = [20, 7, 12]
例如,如果我们在一些整数点处使用CDF,然后在这些CDF值上计算PPF,我们就会得到初始整数
>>> x = np.arange(4) * 2
>>> x
array([0, 2, 4, 6])
>>> prb = hypergeom.cdf(x, M, n, N)
>>> prb
array([ 1.03199174e-04, 5.21155831e-02, 6.08359133e-01,
9.89783282e-01])
>>> hypergeom.ppf(prb, M, n, N)
array([ 0., 2., 4., 6.])
如果我们使用的值不在CDF阶跃函数的结点上,我们将得到下一个更高的整数:
>>> hypergeom.ppf(prb + 1e-8, M, n, N)
array([ 1., 3., 5., 7.])
>>> hypergeom.ppf(prb - 1e-8, M, n, N)
array([ 0., 2., 4., 6.])
拟合分布¶
未冻结分布的主要附加方法与分布参数的估计有关:
- 拟合:分布参数(包括位置)的最大似然估计
和规模
FIT_LOC_SCALE:给定形状参数时位置和比例的估计
nnlf:负对数似然函数
Expect:根据pdf或pmf计算函数的期望值
性能问题和警示说明¶
就速度而言,各个方法的性能因分布和方法的不同而大不相同。方法的结果通过以下两种方式之一获得:通过显式计算,或通过独立于特定分布的通用算法。
一方面,显式计算要求通过解析公式或通过中的特殊函数直接为给定分布指定方法 scipy.special 或 numpy.random 为 rvs 。这些通常是相对较快的计算。
另一方面,如果分布没有指定任何显式计算,则使用泛型方法。要定义分布,只需要pdf或cdf中的一个;所有其他方法都可以使用数值积分和求根得到。但是,这些间接方法可以是 very 慢的。举个例子, rgh = stats.gausshyper.rvs(0.5, 2, 2, 2, size=100) 以一种非常间接的方式创建随机变量,在我的计算机上,100个随机变量大约需要19秒,而来自标准正态分布或t分布的100万个随机变量只需要1秒多一点的时间。
遗留问题¶
中的分发版本 scipy.stats 最近进行了更正和改进,并获得了相当多的测试套件;但是,仍然存在一些问题:
这些分布已经在某些参数范围内进行了测试;但是,在某些拐角范围内,可能会留下一些不正确的结果。
文中的极大似然估计 fit 不适用于所有发行版的默认启动参数,用户需要提供良好的启动参数。此外,对于某些分布,使用最大似然估计器可能天生就不是最佳选择。
构建特定的发行版¶
接下来的示例展示了如何构建您自己的发行版。进一步的例子说明了分布的用法和一些统计检验。
进行连续分布,即进行子类化 rv_continuous¶
进行连续分布是相当简单的。
>>> from scipy import stats
>>> class deterministic_gen(stats.rv_continuous):
... def _cdf(self, x):
... return np.where(x < 0, 0., 1.)
... def _stats(self):
... return 0., 0., 0., 0.
>>> deterministic = deterministic_gen(name="deterministic")
>>> deterministic.cdf(np.arange(-3, 3, 0.5))
array([ 0., 0., 0., 0., 0., 0., 1., 1., 1., 1., 1., 1.])
有趣的是, pdf 现在自动计算:
>>> deterministic.pdf(np.arange(-3, 3, 0.5))
array([ 0.00000000e+00, 0.00000000e+00, 0.00000000e+00,
0.00000000e+00, 0.00000000e+00, 0.00000000e+00,
5.83333333e+04, 4.16333634e-12, 4.16333634e-12,
4.16333634e-12, 4.16333634e-12, 4.16333634e-12])
请注意中提到的性能问题 性能问题和警示说明 。未指定的通用方法的计算可能会变得非常慢,因为只调用一般的方法,而这些方法本质上不能使用有关分布的任何特定信息。因此,作为一个警示例子:
>>> from scipy.integrate import quad
>>> quad(deterministic.pdf, -1e-1, 1e-1)
(4.163336342344337e-13, 0.0)
但是这是不正确的:这个pdf上的积分应该是1。让我们把积分间隔调小一点:
>>> quad(deterministic.pdf, -1e-3, 1e-3) # warning removed
(1.000076872229173, 0.0010625571718182458)
这样看上去好一些。然而,问题源于这样一个事实,即在确定性分布的类别定义中没有指定pdf。
子类化 rv_discrete¶
在以下内容中,我们使用 stats.rv_discrete 以生成离散分布,该离散分布具有以整数为中心的间隔的截断正态的概率。
一般信息
从RV_DISTRIBUTE的文档字符串中, help(stats.rv_discrete) ,
“您可以构造任意离散RV,其中P{X=XK}=PK,方法是(通过VALUES=关键字)将序列元组(XK,PK)传递给RV_DISTRIBUTE初始化方法,该元组仅描述那些以非零概率(PK)出现的X(XK)的值。”
除此之外,此方法还需要一些其他要求才能工作:
关键字 name 是必需的。
分布Xk的支撑点必须是整数。
需要指定有效位数(小数)。
事实上,如果不满足最后两个要求,可能会引发异常,或者结果数字可能不正确。
一个例子
让我们做这项工作吧。首先:
>>> npoints = 20 # number of integer support points of the distribution minus 1
>>> npointsh = npoints // 2
>>> npointsf = float(npoints)
>>> nbound = 4 # bounds for the truncated normal
>>> normbound = (1+1/npointsf) * nbound # actual bounds of truncated normal
>>> grid = np.arange(-npointsh, npointsh+2, 1) # integer grid
>>> gridlimitsnorm = (grid-0.5) / npointsh * nbound # bin limits for the truncnorm
>>> gridlimits = grid - 0.5 # used later in the analysis
>>> grid = grid[:-1]
>>> probs = np.diff(stats.truncnorm.cdf(gridlimitsnorm, -normbound, normbound))
>>> gridint = grid
最后,我们可以子类 rv_discrete :
>>> normdiscrete = stats.rv_discrete(values=(gridint,
... np.round(probs, decimals=7)), name='normdiscrete')
既然我们已经定义了分布,我们就可以访问离散分布的所有常用方法了。
>>> print('mean = %6.4f, variance = %6.4f, skew = %6.4f, kurtosis = %6.4f' %
... normdiscrete.stats(moments='mvsk'))
mean = -0.0000, variance = 6.3302, skew = 0.0000, kurtosis = -0.0076
>>> nd_std = np.sqrt(normdiscrete.stats(moments='v'))
测试实现
让我们随机生成一个样本,并将观察到的频率与概率进行比较。
>>> n_sample = 500
>>> rvs = normdiscrete.rvs(size=n_sample)
>>> f, l = np.histogram(rvs, bins=gridlimits)
>>> sfreq = np.vstack([gridint, f, probs*n_sample]).T
>>> print(sfreq)
[[-1.00000000e+01 0.00000000e+00 2.95019349e-02] # random
[-9.00000000e+00 0.00000000e+00 1.32294142e-01]
[-8.00000000e+00 0.00000000e+00 5.06497902e-01]
[-7.00000000e+00 2.00000000e+00 1.65568919e+00]
[-6.00000000e+00 1.00000000e+00 4.62125309e+00]
[-5.00000000e+00 9.00000000e+00 1.10137298e+01]
[-4.00000000e+00 2.60000000e+01 2.24137683e+01]
[-3.00000000e+00 3.70000000e+01 3.89503370e+01]
[-2.00000000e+00 5.10000000e+01 5.78004747e+01]
[-1.00000000e+00 7.10000000e+01 7.32455414e+01]
[ 0.00000000e+00 7.40000000e+01 7.92618251e+01]
[ 1.00000000e+00 8.90000000e+01 7.32455414e+01]
[ 2.00000000e+00 5.50000000e+01 5.78004747e+01]
[ 3.00000000e+00 5.00000000e+01 3.89503370e+01]
[ 4.00000000e+00 1.70000000e+01 2.24137683e+01]
[ 5.00000000e+00 1.10000000e+01 1.10137298e+01]
[ 6.00000000e+00 4.00000000e+00 4.62125309e+00]
[ 7.00000000e+00 3.00000000e+00 1.65568919e+00]
[ 8.00000000e+00 0.00000000e+00 5.06497902e-01]
[ 9.00000000e+00 0.00000000e+00 1.32294142e-01]
[ 1.00000000e+01 0.00000000e+00 2.95019349e-02]]
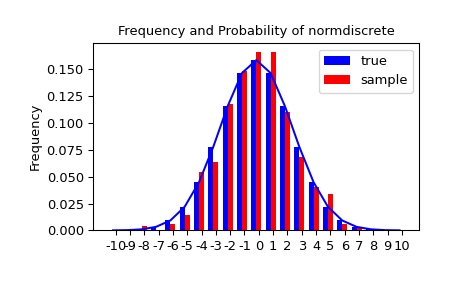
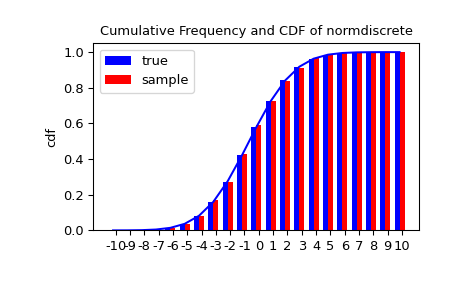
接下来,我们可以测试我们的样本是否由我们的范数离散分布生成。这还验证是否正确生成了随机数。
棋盘检验要求每个仓位中有最少的观测次数。我们把尾箱合并成更大的箱,这样它们就能容纳足够的观测。
>>> f2 = np.hstack([f[:5].sum(), f[5:-5], f[-5:].sum()])
>>> p2 = np.hstack([probs[:5].sum(), probs[5:-5], probs[-5:].sum()])
>>> ch2, pval = stats.chisquare(f2, p2*n_sample)
>>> print('chisquare for normdiscrete: chi2 = %6.3f pvalue = %6.4f' % (ch2, pval))
chisquare for normdiscrete: chi2 = 12.466 pvalue = 0.4090 # random
本例中的p值很高,因此我们可以非常确信我们的随机样本实际上是由分布生成的。
一个样品的分析¶
首先,我们创建一些随机变量。我们设置一个种子,以便在每次运行中都能看到相同的结果。作为示例,我们从学生t分布中选取一个样本:
>>> x = stats.t.rvs(10, size=1000)
这里,我们将t分布所需的形状参数(在统计上与自由度相对应)设置为10。使用size=1000表示我们的样本由1000个独立绘制的(伪)随机数组成。因为我们没有指定关键字参数 loc 和 scale ,则将它们设置为默认值0和1。
描述性统计¶
x 是Numpy数组,并且我们可以直接访问所有数组方法,例如,
>>> print(x.min()) # equivalent to np.min(x)
-3.78975572422 # random
>>> print(x.max()) # equivalent to np.max(x)
5.26327732981 # random
>>> print(x.mean()) # equivalent to np.mean(x)
0.0140610663985 # random
>>> print(x.var()) # equivalent to np.var(x))
1.28899386208 # random
样品的性质与其理论性质相比如何?
>>> m, v, s, k = stats.t.stats(10, moments='mvsk')
>>> n, (smin, smax), sm, sv, ss, sk = stats.describe(x)
>>> sstr = '%-14s mean = %6.4f, variance = %6.4f, skew = %6.4f, kurtosis = %6.4f'
>>> print(sstr % ('distribution:', m, v, s ,k))
distribution: mean = 0.0000, variance = 1.2500, skew = 0.0000, kurtosis = 1.0000 # random
>>> print(sstr % ('sample:', sm, sv, ss, sk))
sample: mean = 0.0141, variance = 1.2903, skew = 0.2165, kurtosis = 1.0556 # random
注: stats.describe 对方差使用无偏估计器,而np.var是有偏估计器。
对于我们的样本,样本统计与理论统计相差很小。
t检验和KS检验¶
我们可以使用t检验来检验我们样本的平均值是否在统计上显著不同于理论预期。
>>> print('t-statistic = %6.3f pvalue = %6.4f' % stats.ttest_1samp(x, m))
t-statistic = 0.391 pvalue = 0.6955 # random
p值是0.7,这意味着在α误差为例如10%的情况下,我们不能拒绝样本均值等于零的假设,即标准t分布的期望值。
作为练习,我们还可以直接计算我们的Ttest,而不使用所提供的函数,这应该会给我们相同的答案,它确实是这样做的:
>>> tt = (sm-m)/np.sqrt(sv/float(n)) # t-statistic for mean
>>> pval = stats.t.sf(np.abs(tt), n-1)*2 # two-sided pvalue = Prob(abs(t)>tt)
>>> print('t-statistic = %6.3f pvalue = %6.4f' % (tt, pval))
t-statistic = 0.391 pvalue = 0.6955 # random
Kolmogorov-Smirnov检验可以用来检验样本来自标准t分布的假设。
>>> print('KS-statistic D = %6.3f pvalue = %6.4f' % stats.kstest(x, 't', (10,)))
KS-statistic D = 0.016 pvalue = 0.9571 # random
同样,p值足够高,以至于我们不能拒绝随机样本确实按照t分布分布的假设。在实际应用中,我们不知道底层分布是什么。如果我们针对标准正态分布对我们的样本执行Kolmogorov-Smirnov检验,那么我们也不能拒绝假设我们的样本是由正态分布产生的,因为在本例中,p值几乎是40%。
>>> print('KS-statistic D = %6.3f pvalue = %6.4f' % stats.kstest(x, 'norm'))
KS-statistic D = 0.028 pvalue = 0.3918 # random
然而,标准正态分布的方差为1,而我们的样本的方差为1.29。如果我们标准化我们的样本,并根据正态分布对其进行测试,那么p值再次足够大,以至于我们不能拒绝样本来自正态分布的假设。
>>> d, pval = stats.kstest((x-x.mean())/x.std(), 'norm')
>>> print('KS-statistic D = %6.3f pvalue = %6.4f' % (d, pval))
KS-statistic D = 0.032 pvalue = 0.2397 # random
注:Kolmogorov-Smirnov检验假设我们针对给定参数的分布进行测试,因为在最后一种情况下,我们估计了均值和方差,违反了这一假设,并且p值所基于的测试统计量的分布是不正确的。
分布的尾部¶
最后,我们可以检查分布的上尾。我们可以使用百分点函数PPF(CDF函数的逆函数)来获得临界值,或者,更直接地,我们可以使用生存函数的逆函数
>>> crit01, crit05, crit10 = stats.t.ppf([1-0.01, 1-0.05, 1-0.10], 10)
>>> print('critical values from ppf at 1%%, 5%% and 10%% %8.4f %8.4f %8.4f' % (crit01, crit05, crit10))
critical values from ppf at 1%, 5% and 10% 2.7638 1.8125 1.3722
>>> print('critical values from isf at 1%%, 5%% and 10%% %8.4f %8.4f %8.4f' % tuple(stats.t.isf([0.01,0.05,0.10],10)))
critical values from isf at 1%, 5% and 10% 2.7638 1.8125 1.3722
>>> freq01 = np.sum(x>crit01) / float(n) * 100
>>> freq05 = np.sum(x>crit05) / float(n) * 100
>>> freq10 = np.sum(x>crit10) / float(n) * 100
>>> print('sample %%-frequency at 1%%, 5%% and 10%% tail %8.4f %8.4f %8.4f' % (freq01, freq05, freq10))
sample %-frequency at 1%, 5% and 10% tail 1.4000 5.8000 10.5000 # random
在所有三种情况下,我们的样本在顶部尾部的权重都大于底层分布。我们可以简单地检查一下更大的样本,看看是否有更接近的匹配。在这种情况下,经验频率与理论概率相当接近,但如果我们重复几次,波动仍然相当大。
>>> freq05l = np.sum(stats.t.rvs(10, size=10000) > crit05) / 10000.0 * 100
>>> print('larger sample %%-frequency at 5%% tail %8.4f' % freq05l)
larger sample %-frequency at 5% tail 4.8000 # random
我们还可以将其与正态分布的尾部进行比较,后者在尾部的权重较小:
>>> print('tail prob. of normal at 1%%, 5%% and 10%% %8.4f %8.4f %8.4f' %
... tuple(stats.norm.sf([crit01, crit05, crit10])*100))
tail prob. of normal at 1%, 5% and 10% 0.2857 3.4957 8.5003
平方检验可用于测试对于有限数量的箱,观察到的频率是否与假设分布的概率显著不同。
>>> quantiles = [0.0, 0.01, 0.05, 0.1, 1-0.10, 1-0.05, 1-0.01, 1.0]
>>> crit = stats.t.ppf(quantiles, 10)
>>> crit
array([ -inf, -2.76376946, -1.81246112, -1.37218364, 1.37218364,
1.81246112, 2.76376946, inf])
>>> n_sample = x.size
>>> freqcount = np.histogram(x, bins=crit)[0]
>>> tprob = np.diff(quantiles)
>>> nprob = np.diff(stats.norm.cdf(crit))
>>> tch, tpval = stats.chisquare(freqcount, tprob*n_sample)
>>> nch, npval = stats.chisquare(freqcount, nprob*n_sample)
>>> print('chisquare for t: chi2 = %6.2f pvalue = %6.4f' % (tch, tpval))
chisquare for t: chi2 = 2.30 pvalue = 0.8901 # random
>>> print('chisquare for normal: chi2 = %6.2f pvalue = %6.4f' % (nch, npval))
chisquare for normal: chi2 = 64.60 pvalue = 0.0000 # random
我们看到,标准正态分布显然是被拒绝的,而标准t-分布是不能被拒绝的。由于我们样本的方差不同于这两个标准分布,我们可以再次进行测试,同时考虑到对规模和位置的估计。
分布的拟合方法可以用来估计分布的参数,并利用估计的分布的概率重复检验。
>>> tdof, tloc, tscale = stats.t.fit(x)
>>> nloc, nscale = stats.norm.fit(x)
>>> tprob = np.diff(stats.t.cdf(crit, tdof, loc=tloc, scale=tscale))
>>> nprob = np.diff(stats.norm.cdf(crit, loc=nloc, scale=nscale))
>>> tch, tpval = stats.chisquare(freqcount, tprob*n_sample)
>>> nch, npval = stats.chisquare(freqcount, nprob*n_sample)
>>> print('chisquare for t: chi2 = %6.2f pvalue = %6.4f' % (tch, tpval))
chisquare for t: chi2 = 1.58 pvalue = 0.9542 # random
>>> print('chisquare for normal: chi2 = %6.2f pvalue = %6.4f' % (nch, npval))
chisquare for normal: chi2 = 11.08 pvalue = 0.0858 # random
考虑到估计的参数,我们仍然可以拒绝假设我们的样本来自正态分布(在5%的水平上),但同样,在p值为0.95的情况下,我们不能拒绝t分布。
正态分布的特殊检验¶
由于正态分布是统计中最常见的分布,因此可以使用几个附加函数来测试样本是否可以从正态分布中提取。
首先,我们可以检验样本的偏度和峰度是否与正态分布的偏差和峰度有显著差异:
>>> print('normal skewtest teststat = %6.3f pvalue = %6.4f' % stats.skewtest(x))
normal skewtest teststat = 2.785 pvalue = 0.0054 # random
>>> print('normal kurtosistest teststat = %6.3f pvalue = %6.4f' % stats.kurtosistest(x))
normal kurtosistest teststat = 4.757 pvalue = 0.0000 # random
这两个检验在正态检验中结合起来。
>>> print('normaltest teststat = %6.3f pvalue = %6.4f' % stats.normaltest(x))
normaltest teststat = 30.379 pvalue = 0.0000 # random
在所有三个检验中,p值都很低,我们可以拒绝我们的样本具有正态分布的偏态和峰度的假设。
因为我们样本的偏度和峰度是基于中心矩的,所以如果我们测试标准化样本,我们会得到完全相同的结果:
>>> print('normaltest teststat = %6.3f pvalue = %6.4f' %
... stats.normaltest((x-x.mean())/x.std()))
normaltest teststat = 30.379 pvalue = 0.0000 # random
因为正态被如此强烈地拒绝,我们可以检查正态检验对于其他情况是否给出合理的结果:
>>> print('normaltest teststat = %6.3f pvalue = %6.4f' %
... stats.normaltest(stats.t.rvs(10, size=100)))
normaltest teststat = 4.698 pvalue = 0.0955 # random
>>> print('normaltest teststat = %6.3f pvalue = %6.4f' %
... stats.normaltest(stats.norm.rvs(size=1000)))
normaltest teststat = 0.613 pvalue = 0.7361 # random
当检验t分布观测值的小样本和正态分布观测值的大样本的正态性时,无论哪种情况,我们都不能拒绝样本来自正态分布的零假设。在第一种情况下,这是因为检验不足以区分小样本中的t和正态分布随机变量。
比较两个样本¶
下面给我们两个样本,这两个样本既可以来自相同的分布,也可以来自不同的分布,我们想要检验这些样本是否具有相同的统计特性。
比较手段¶
用相同的方法测试样品:
>>> rvs1 = stats.norm.rvs(loc=5, scale=10, size=500)
>>> rvs2 = stats.norm.rvs(loc=5, scale=10, size=500)
>>> stats.ttest_ind(rvs1, rvs2)
Ttest_indResult(statistic=-0.5489036175088705, pvalue=0.5831943748663959) # random
用不同的方法对样品进行测试:
>>> rvs3 = stats.norm.rvs(loc=8, scale=10, size=500)
>>> stats.ttest_ind(rvs1, rvs3)
Ttest_indResult(statistic=-4.533414290175026, pvalue=6.507128186389019e-06) # random
两样本Ks2样本的Kolmogorov-Smirnov检验¶
对于两个样本都来自同一分布的示例,我们不能拒绝零假设,因为p值很高
>>> stats.ks_2samp(rvs1, rvs2)
KstestResult(statistic=0.026, pvalue=0.9959527565364388) # random
在第二个示例中,由于p值低于1%,因此对于不同的位置(即,均值),我们可以拒绝零假设
>>> stats.ks_2samp(rvs1, rvs3)
KstestResult(statistic=0.114, pvalue=0.00299005061044668) # random
核密度估计¶
统计学中的一项常见任务是从一组数据样本中估计随机变量的概率密度函数(PDF)。这项任务称为密度估计。最广为人知的工具是直方图。直方图是可视化的有用工具(主要是因为每个人都能理解它),但是不能非常有效地利用可用的数据。对于同样的任务,核密度估计(KDE)是一种更有效的工具。这个 gaussian_kde 估计器既可用于估计单变量数据的PDF,也可用于估计多变量数据的PDF。如果数据是单峰的,则效果最好。
单变量估计¶
我们从最少量的数据开始,以便了解如何 gaussian_kde 工作方式以及带宽选择的不同选项的作用。从PDF中采样的数据在图的底部显示为蓝色虚线(这称为地毯图):
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> x1 = np.array([-7, -5, 1, 4, 5], dtype=np.float64)
>>> kde1 = stats.gaussian_kde(x1)
>>> kde2 = stats.gaussian_kde(x1, bw_method='silverman')
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> ax.plot(x1, np.zeros(x1.shape), 'b+', ms=20) # rug plot
>>> x_eval = np.linspace(-10, 10, num=200)
>>> ax.plot(x_eval, kde1(x_eval), 'k-', label="Scott's Rule")
>>> ax.plot(x_eval, kde2(x_eval), 'r-', label="Silverman's Rule")
>>> plt.show()
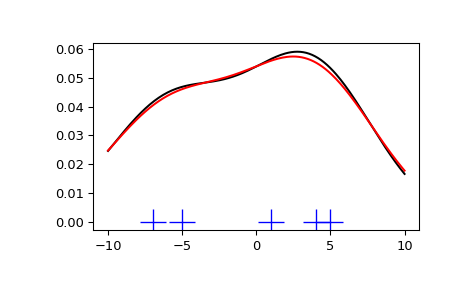
我们看到,斯科特规则和西尔弗曼规则之间的差别很小,并且有限数据量的带宽选择可能有点太宽了。我们可以定义我们自己的带宽函数,以得到较不平滑的结果。
>>> def my_kde_bandwidth(obj, fac=1./5):
... """We use Scott's Rule, multiplied by a constant factor."""
... return np.power(obj.n, -1./(obj.d+4)) * fac
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> ax.plot(x1, np.zeros(x1.shape), 'b+', ms=20) # rug plot
>>> kde3 = stats.gaussian_kde(x1, bw_method=my_kde_bandwidth)
>>> ax.plot(x_eval, kde3(x_eval), 'g-', label="With smaller BW")
>>> plt.show()
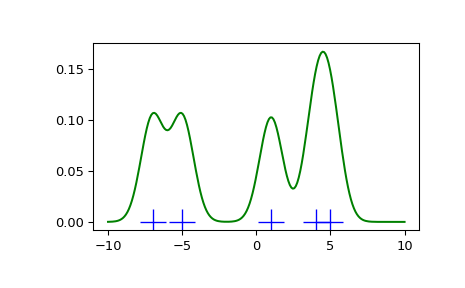
我们看到,如果我们将带宽设置得非常窄,则获得的概率密度函数(PDF)的估计简单地是每个数据点周围的高斯和。
现在,我们举一个更实际的例子,看看两个可用带宽选择规则之间的区别。众所周知,这些规则适用于(接近)正态分布,但即使对于非常强烈的非正态分布的单峰分布,它们也相当有效。作为非正态分布,我们采用5个自由度的学生T分布。
import numpy as np
import matplotlib.pyplot as plt
from scipy import stats
rng = np.random.default_rng()
x1 = rng.normal(size=200) # random data, normal distribution
xs = np.linspace(x1.min()-1, x1.max()+1, 200)
kde1 = stats.gaussian_kde(x1)
kde2 = stats.gaussian_kde(x1, bw_method='silverman')
fig = plt.figure(figsize=(8, 6))
ax1 = fig.add_subplot(211)
ax1.plot(x1, np.zeros(x1.shape), 'b+', ms=12) # rug plot
ax1.plot(xs, kde1(xs), 'k-', label="Scott's Rule")
ax1.plot(xs, kde2(xs), 'b-', label="Silverman's Rule")
ax1.plot(xs, stats.norm.pdf(xs), 'r--', label="True PDF")
ax1.set_xlabel('x')
ax1.set_ylabel('Density')
ax1.set_title("Normal (top) and Student's T$_{df=5}$ (bottom) distributions")
ax1.legend(loc=1)
x2 = stats.t.rvs(5, size=200, random_state=rng) # random data, T distribution
xs = np.linspace(x2.min() - 1, x2.max() + 1, 200)
kde3 = stats.gaussian_kde(x2)
kde4 = stats.gaussian_kde(x2, bw_method='silverman')
ax2 = fig.add_subplot(212)
ax2.plot(x2, np.zeros(x2.shape), 'b+', ms=12) # rug plot
ax2.plot(xs, kde3(xs), 'k-', label="Scott's Rule")
ax2.plot(xs, kde4(xs), 'b-', label="Silverman's Rule")
ax2.plot(xs, stats.t.pdf(xs, 5), 'r--', label="True PDF")
ax2.set_xlabel('x')
ax2.set_ylabel('Density')
plt.show()
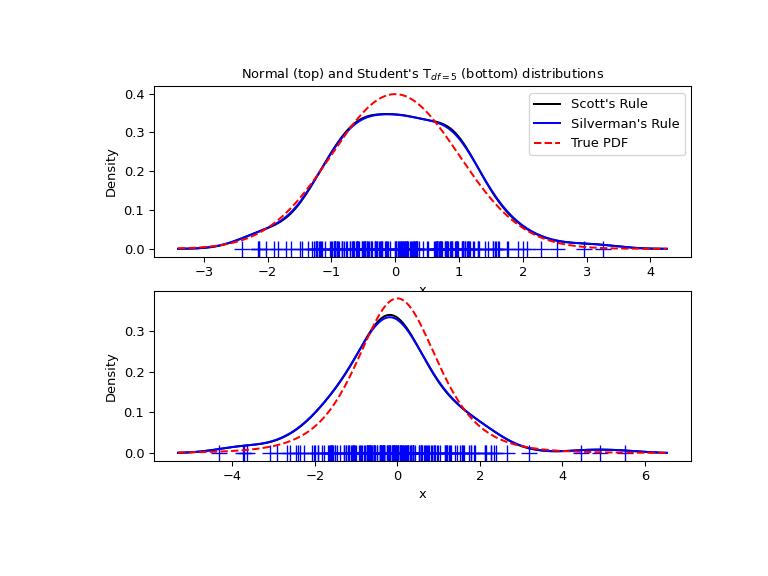
我们现在看一看具有一个更宽的和一个更窄的高斯特征的双峰分布。我们预计这将是一个更难近似的密度,因为精确解析每个特征所需的带宽不同。
>>> from functools import partial
>>> loc1, scale1, size1 = (-2, 1, 175)
>>> loc2, scale2, size2 = (2, 0.2, 50)
>>> x2 = np.concatenate([np.random.normal(loc=loc1, scale=scale1, size=size1),
... np.random.normal(loc=loc2, scale=scale2, size=size2)])
>>> x_eval = np.linspace(x2.min() - 1, x2.max() + 1, 500)
>>> kde = stats.gaussian_kde(x2)
>>> kde2 = stats.gaussian_kde(x2, bw_method='silverman')
>>> kde3 = stats.gaussian_kde(x2, bw_method=partial(my_kde_bandwidth, fac=0.2))
>>> kde4 = stats.gaussian_kde(x2, bw_method=partial(my_kde_bandwidth, fac=0.5))
>>> pdf = stats.norm.pdf
>>> bimodal_pdf = pdf(x_eval, loc=loc1, scale=scale1) * float(size1) / x2.size + \
... pdf(x_eval, loc=loc2, scale=scale2) * float(size2) / x2.size
>>> fig = plt.figure(figsize=(8, 6))
>>> ax = fig.add_subplot(111)
>>> ax.plot(x2, np.zeros(x2.shape), 'b+', ms=12)
>>> ax.plot(x_eval, kde(x_eval), 'k-', label="Scott's Rule")
>>> ax.plot(x_eval, kde2(x_eval), 'b-', label="Silverman's Rule")
>>> ax.plot(x_eval, kde3(x_eval), 'g-', label="Scott * 0.2")
>>> ax.plot(x_eval, kde4(x_eval), 'c-', label="Scott * 0.5")
>>> ax.plot(x_eval, bimodal_pdf, 'r--', label="Actual PDF")
>>> ax.set_xlim([x_eval.min(), x_eval.max()])
>>> ax.legend(loc=2)
>>> ax.set_xlabel('x')
>>> ax.set_ylabel('Density')
>>> plt.show()
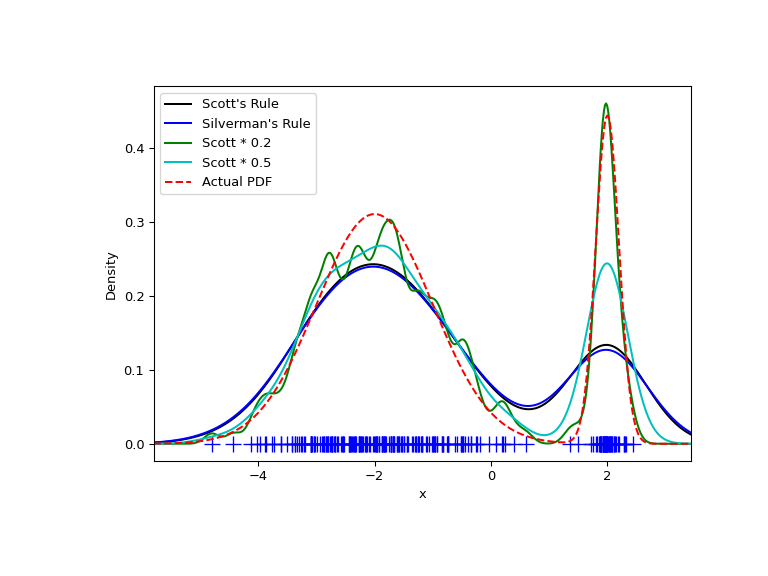
正如预期的那样,由于双峰分布的两个特性的特征大小不同,KDE并不像我们希望的那样接近真正的PDF。通过将默认带宽减半 (Scott * 0.5 ),我们可以做得更好一些,而使用比默认带宽小5倍的带宽不够平滑。不过,在这种情况下,我们真正需要的是非均匀(自适应)带宽。
多变量估计¶
使用 gaussian_kde 我们可以进行多变量估计,也可以进行单变量估计。我们证明了二元情形。首先,我们用两个变量相关的模型产生一些随机数据。
>>> def measure(n):
... """Measurement model, return two coupled measurements."""
... m1 = np.random.normal(size=n)
... m2 = np.random.normal(scale=0.5, size=n)
... return m1+m2, m1-m2
>>> m1, m2 = measure(2000)
>>> xmin = m1.min()
>>> xmax = m1.max()
>>> ymin = m2.min()
>>> ymax = m2.max()
然后,我们将KDE应用于数据:
>>> X, Y = np.mgrid[xmin:xmax:100j, ymin:ymax:100j]
>>> positions = np.vstack([X.ravel(), Y.ravel()])
>>> values = np.vstack([m1, m2])
>>> kernel = stats.gaussian_kde(values)
>>> Z = np.reshape(kernel.evaluate(positions).T, X.shape)
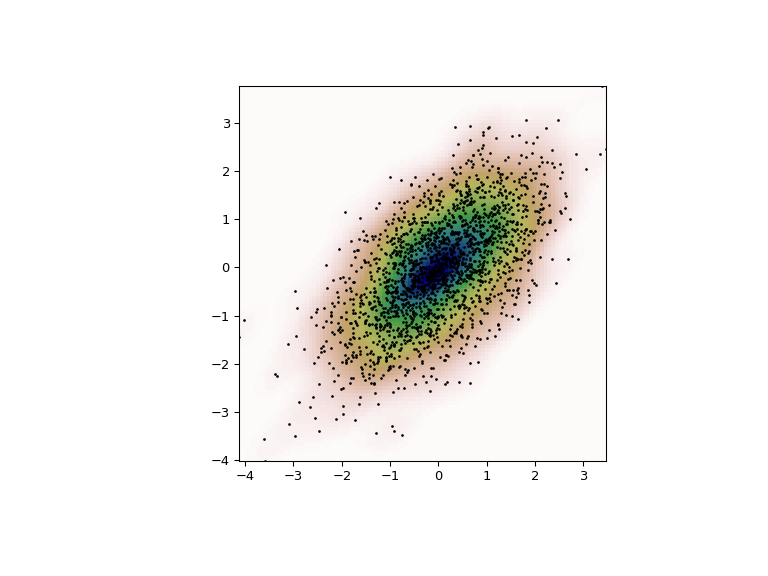
最后,我们将估计的双变量分布绘制为颜色图,并将各个数据点绘制在顶部。
>>> fig = plt.figure(figsize=(8, 6))
>>> ax = fig.add_subplot(111)
>>> ax.imshow(np.rot90(Z), cmap=plt.cm.gist_earth_r,
... extent=[xmin, xmax, ymin, ymax])
>>> ax.plot(m1, m2, 'k.', markersize=2)
>>> ax.set_xlim([xmin, xmax])
>>> ax.set_ylim([ymin, ymax])
>>> plt.show()
多尺度图相关(MGC)¶
使用 multiscale_graphcorr ,我们可以对高维和非线性数据进行独立性检验。在开始之前,我们先导入一些有用的包:
>>> import numpy as np
>>> import matplotlib.pyplot as plt; plt.style.use('classic')
>>> from scipy.stats import multiscale_graphcorr
让我们使用一个自定义绘图函数来绘制数据关系:
>>> def mgc_plot(x, y, sim_name, mgc_dict=None, only_viz=False,
... only_mgc=False):
... """Plot sim and MGC-plot"""
... if not only_mgc:
... # simulation
... plt.figure(figsize=(8, 8))
... ax = plt.gca()
... ax.set_title(sim_name + " Simulation", fontsize=20)
... ax.scatter(x, y)
... ax.set_xlabel('X', fontsize=15)
... ax.set_ylabel('Y', fontsize=15)
... ax.axis('equal')
... ax.tick_params(axis="x", labelsize=15)
... ax.tick_params(axis="y", labelsize=15)
... plt.show()
... if not only_viz:
... # local correlation map
... plt.figure(figsize=(8,8))
... ax = plt.gca()
... mgc_map = mgc_dict["mgc_map"]
... # draw heatmap
... ax.set_title("Local Correlation Map", fontsize=20)
... im = ax.imshow(mgc_map, cmap='YlGnBu')
... # colorbar
... cbar = ax.figure.colorbar(im, ax=ax)
... cbar.ax.set_ylabel("", rotation=-90, va="bottom")
... ax.invert_yaxis()
... # Turn spines off and create white grid.
... for edge, spine in ax.spines.items():
... spine.set_visible(False)
... # optimal scale
... opt_scale = mgc_dict["opt_scale"]
... ax.scatter(opt_scale[0], opt_scale[1],
... marker='X', s=200, color='red')
... # other formatting
... ax.tick_params(bottom="off", left="off")
... ax.set_xlabel('#Neighbors for X', fontsize=15)
... ax.set_ylabel('#Neighbors for Y', fontsize=15)
... ax.tick_params(axis="x", labelsize=15)
... ax.tick_params(axis="y", labelsize=15)
... ax.set_xlim(0, 100)
... ax.set_ylim(0, 100)
... plt.show()
我们先来看一些线性数据:
>>> rng = np.random.default_rng()
>>> x = np.linspace(-1, 1, num=100)
>>> y = x + 0.3 * rng.random(x.size)
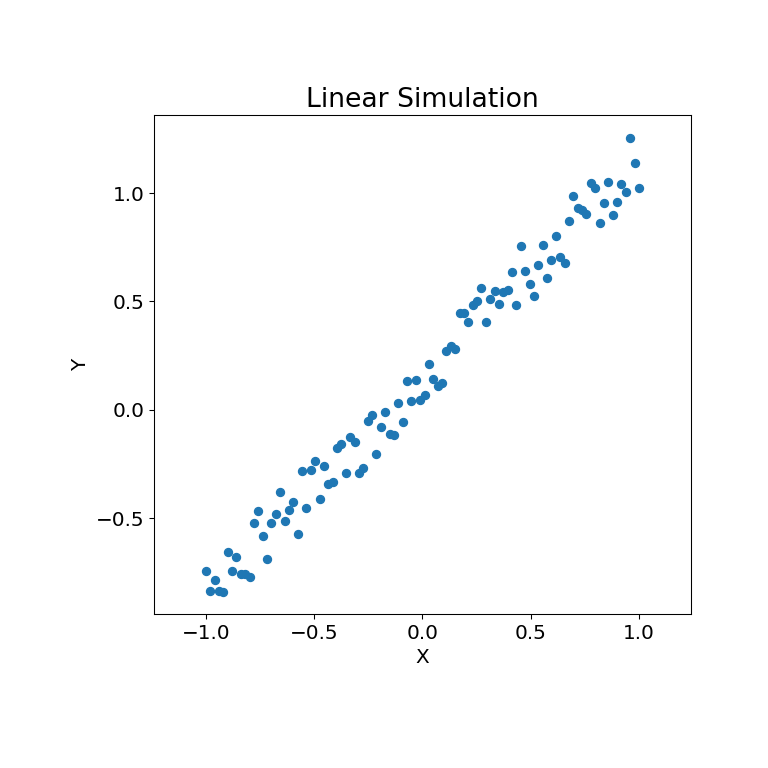
仿真关系如下图所示:
>>> mgc_plot(x, y, "Linear", only_viz=True)
现在,我们可以看到下面可视化的测试统计数据、p值和MGC图。最佳比例在地图上显示为红色“x”:
>>> stat, pvalue, mgc_dict = multiscale_graphcorr(x, y)
>>> print("MGC test statistic: ", round(stat, 1))
MGC test statistic: 1.0
>>> print("P-value: ", round(pvalue, 1))
P-value: 0.0
>>> mgc_plot(x, y, "Linear", mgc_dict, only_mgc=True)
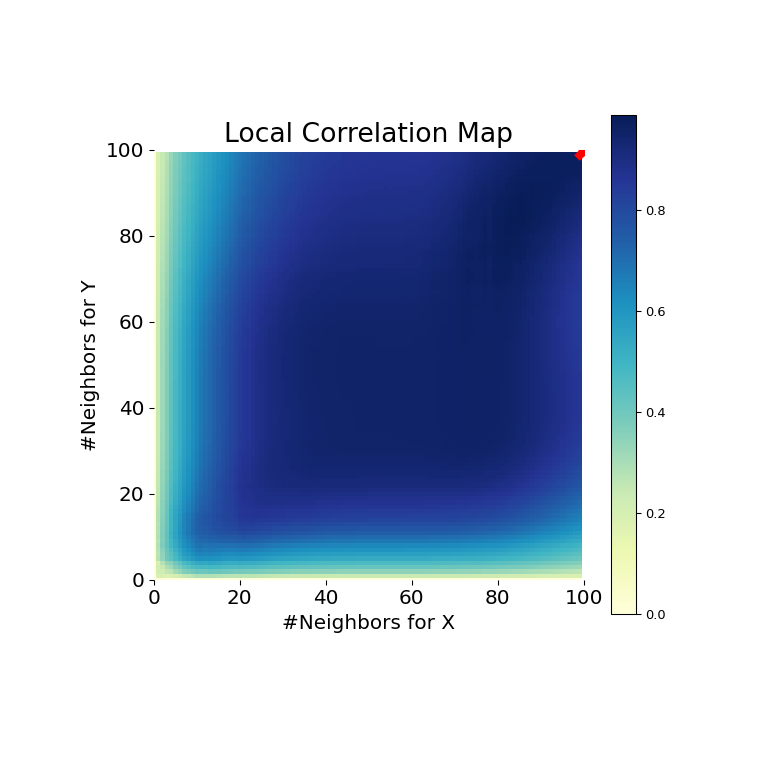
从这里可以清楚地看出,MGC能够确定输入数据矩阵之间关系,因为p值非常低,而MGC测试统计量相对较高。MGC-MAP指示 强线性关系 。直观地说,这是因为拥有更多的邻居将有助于识别 \(x\) 和 \(y\) 。这种情况下的最佳比例是 相当于全球尺度 ,在地图上标有一个红点。
对于非线性数据集也可以这样做。以下内容 \(x\) 和 \(y\) 阵列源自非线性模拟:
>>> unif = np.array(rng.uniform(0, 5, size=100))
>>> x = unif * np.cos(np.pi * unif)
>>> y = unif * np.sin(np.pi * unif) + 0.4 * rng.random(x.size)
仿真关系如下图所示:
>>> mgc_plot(x, y, "Spiral", only_viz=True)
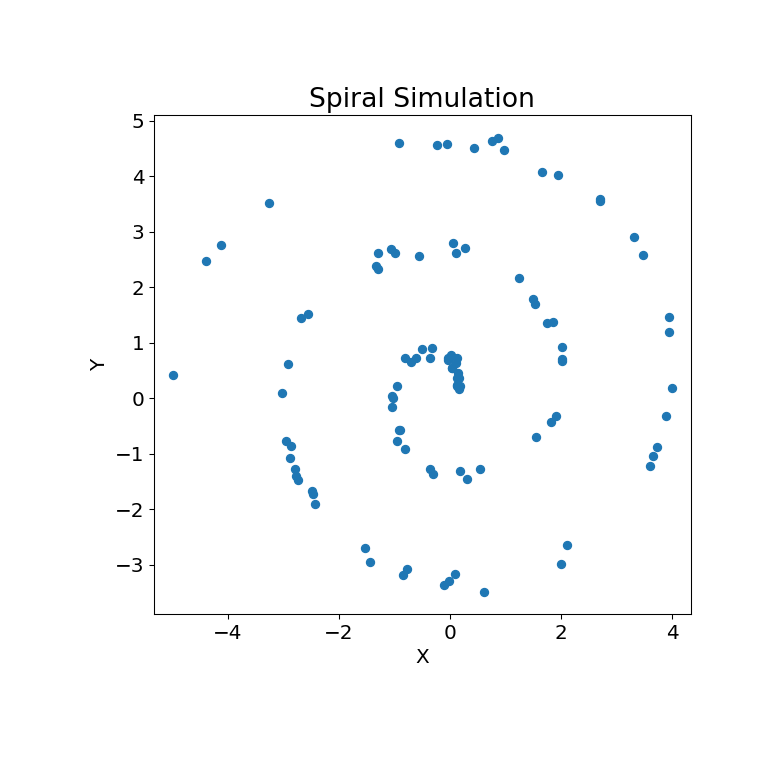
现在,我们可以看到下面可视化的测试统计数据、p值和MGC图。最佳比例在地图上显示为红色“x”:
>>> stat, pvalue, mgc_dict = multiscale_graphcorr(x, y)
>>> print("MGC test statistic: ", round(stat, 1))
MGC test statistic: 0.2 # random
>>> print("P-value: ", round(pvalue, 1))
P-value: 0.0
>>> mgc_plot(x, y, "Spiral", mgc_dict, only_mgc=True)
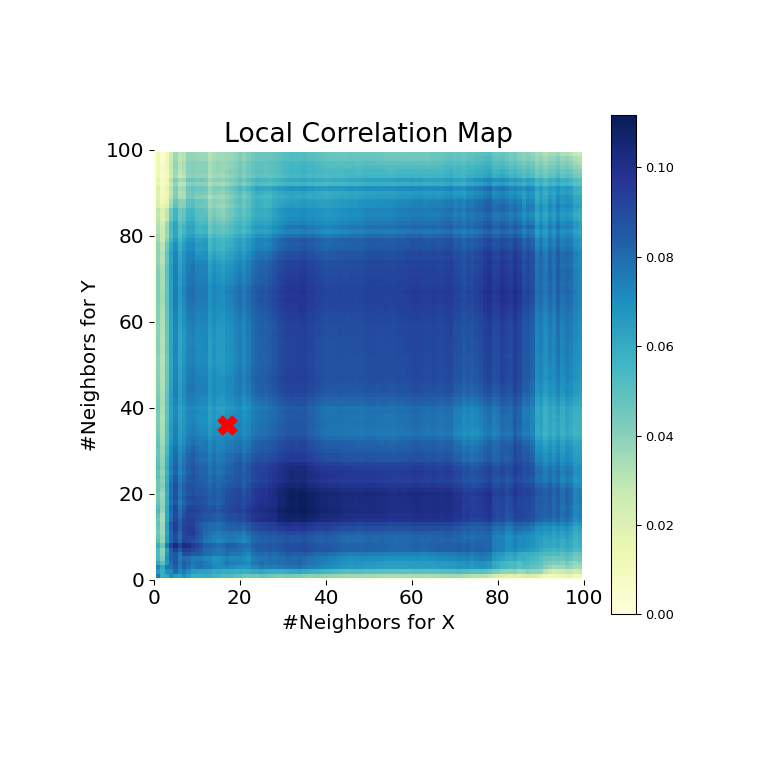
从这里可以清楚地看出,MGC能够再次确定关系,因为p值非常低,而MGC测试统计量相对较高。MGC-MAP指示 强非线性关系 。这种情况下的最佳比例是 相当于当地的比例尺 ,在地图上标有一个红点。
拟蒙特卡罗¶
在讨论准蒙特卡罗(QMC)之前,先简要介绍一下蒙特卡罗(MC)。MC方法,或MC实验,是一类广泛的计算算法,它依赖于重复随机抽样来获得数值结果。其基本概念是使用随机性来解决原则上可能是确定性的问题。它们经常用于物理和数学问题,并且在难以或不可能使用其他方法时最有用。MC方法主要用于三类问题:最优化、数值积分和根据概率分布生成图纸。
生成具有特定属性的随机数是一个比听起来更复杂的问题。简单的MC方法用于采样点的独立同分布(IID)。但是,生成多组随机点可能会产生截然不同的结果。
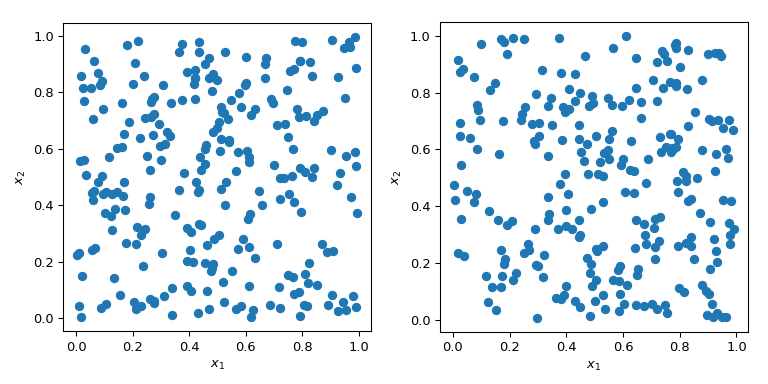
在上图中的这两种情况下,点都是随机生成的,而不知道以前绘制的点。很明显,空间的一些区域没有被探索-这可能会在模拟中造成问题,因为一组特定的点可能会触发完全不同的行为。
MC的一个很大的优点是它具有已知的收敛性。让我们看一下5个维度的平方和的平均值:
使用 \(x_j \sim \mathcal{{U}}(0,1)\) 。它有一个已知的平均值, \(\mu = 5/3+5(5-1)/4\) 。使用MC抽样,我们可以用数值计算该平均值,并且近似误差遵循理论上的 \(O(n^{{-1/2}})\) 。
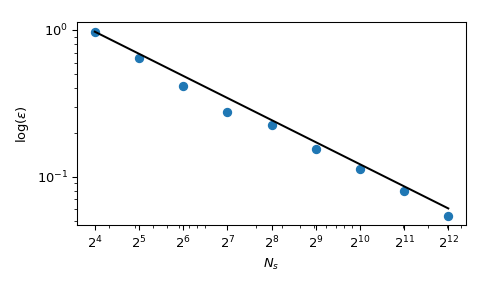
虽然收敛是有保证的,但实践者往往希望有一个更具确定性的探索过程。对于正常的MC,种子可以用来进行可重复的过程。但是修复种子会破坏收敛特性:给定的种子可能适用于给定的一类问题,而不适用于另一类问题。
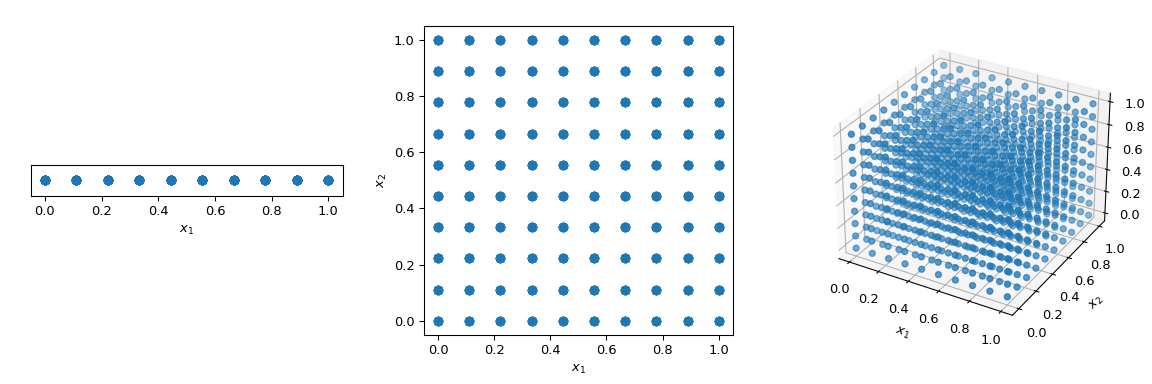
要以确定性的方式在空间中漫步,通常要使用跨越所有参数维度的规则网格,也称为饱和设计。让我们考虑一下单位超立方体,所有边界都在0到1之间。现在,如果点之间的距离为0.1,填充单位间隔所需的点数将是10。在二维超立方体中,相同的间距需要100个点,而在三维空间中,需要1000个点。随着维数的增加,填充空间所需的实验数量随着空间维数的增加呈指数级增加。这种指数增长被称为“维度诅咒”。
>>> import numpy as np
>>> disc = 10
>>> x1 = np.linspace(0, 1, disc)
>>> x2 = np.linspace(0, 1, disc)
>>> x3 = np.linspace(0, 1, disc)
>>> x1, x2, x3 = np.meshgrid(x1, x2, x3)
为了缓解这个问题,设计了QMC方法。它们是确定性的,具有很好的空间覆盖性,其中一些可以继续下去,并保持良好的性质。与MC方法的主要区别在于,这些点不是IID,但它们知道以前的点。因此,有些方法也称为序列。
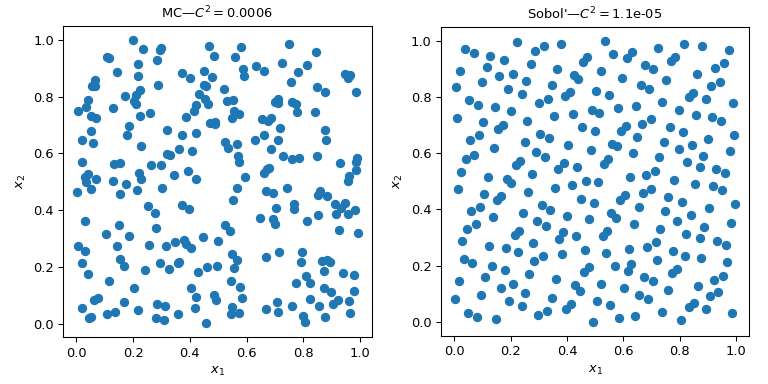
此图显示2组256个点。左边的设计是纯MC设计,而右边的设计是QMC设计,使用 苏博尔的 方法。我们清楚地看到,QMC版本更加统一。点在边界附近采样较好,簇或间隙较少。
评估一致性的一种方法是使用一种称为差异的测量方法。在这里,不一致之处在于 苏博尔的 积分优于粗MC。
回到均值的计算,QMC方法对于误差也有较好的收敛速度。他们可以实现 \(O(n^{{-1}})\) 对于这个函数,在非常光滑的函数上的比率甚至更高。此图显示, 苏博尔的 方法的速率为 \(O(n^{{-1}})\) :
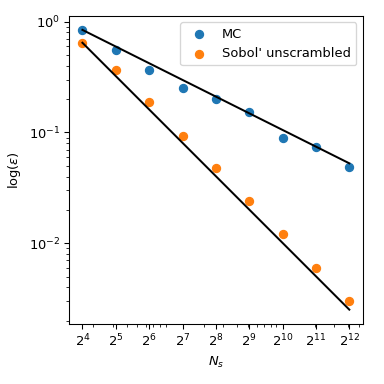
我们指的是 scipy.stats.qmc 了解更多数学细节。
计算差额¶
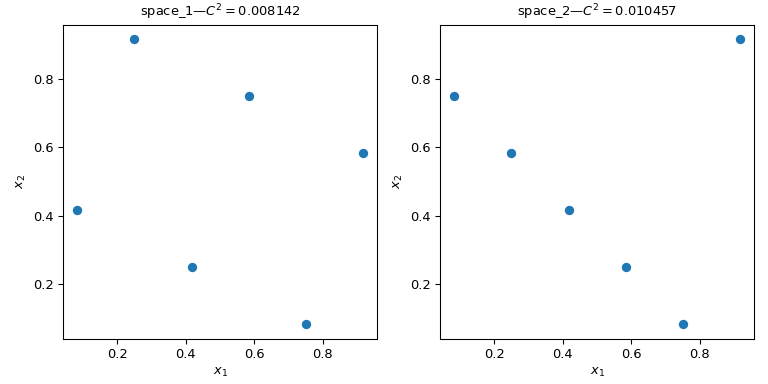
让我们考虑两组观点。从下图可以清楚地看出,左边的设计比右边的设计覆盖了更多的空间。这可以使用 discrepancy 测量。差异越小,样本越均匀。
>>> import numpy as np
>>> from scipy.stats import qmc
>>> space_1 = np.array([[1, 3], [2, 6], [3, 2], [4, 5], [5, 1], [6, 4]])
>>> space_2 = np.array([[1, 5], [2, 4], [3, 3], [4, 2], [5, 1], [6, 6]])
>>> l_bounds = [0.5, 0.5]
>>> u_bounds = [6.5, 6.5]
>>> space_1 = qmc.scale(space_1, l_bounds, u_bounds, reverse=True)
>>> space_2 = qmc.scale(space_2, l_bounds, u_bounds, reverse=True)
>>> qmc.discrepancy(space_1)
0.008142039609053464
>>> qmc.discrepancy(space_2)
0.010456854423869011
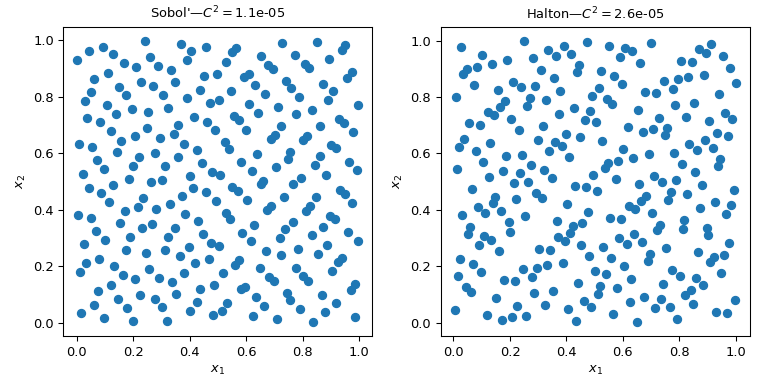
使用QMC引擎¶
实现了几个QMC采样器/引擎。下面我们来看两个最常用的QMC方法: Sobol 和 Halton 序列。
"""Sobol' and Halton sequences."""
from scipy.stats import qmc
import numpy as np
import matplotlib.pyplot as plt
rng = np.random.default_rng()
n_sample = 256
dim = 2
sample = {}
# Sobol'
engine = qmc.Sobol(d=dim, seed=rng)
sample["Sobol'"] = engine.random(n_sample)
# Halton
engine = qmc.Halton(d=dim, seed=rng)
sample["Halton"] = engine.random(n_sample)
fig, axs = plt.subplots(1, 2, figsize=(8, 4))
for i, kind in enumerate(sample):
axs[i].scatter(sample[kind][:, 0], sample[kind][:, 1])
axs[i].set_aspect('equal')
axs[i].set_xlabel(r'$x_1$')
axs[i].set_ylabel(r'$x_2$')
axs[i].set_title(f'{kind}—$C^2 = ${qmc.discrepancy(sample[kind]):.2}')
plt.tight_layout()
plt.show()
警告
QMC方法需要特别小心,用户必须阅读文档以避免常见陷阱。 苏博尔的 例如,需要2的幂之后的多个点。此外,细化、烧录或其他点选择可能会破坏序列的属性,并产生一组不会比MC更好的点。
QMC引擎是状态感知的。这意味着您可以继续该序列、跳过某些点或将其重置。让我们从 Halton 。然后要求第二组5分:
>>> from scipy.stats import qmc
>>> engine = qmc.Halton(d=2)
>>> engine.random(5)
array([[0.22166437, 0.07980522], # random
[0.72166437, 0.93165708],
[0.47166437, 0.41313856],
[0.97166437, 0.19091633],
[0.01853937, 0.74647189]])
>>> engine.random(5)
array([[0.51853937, 0.52424967], # random
[0.26853937, 0.30202745],
[0.76853937, 0.857583 ],
[0.14353937, 0.63536078],
[0.64353937, 0.01807683]])
现在我们重新设置序列。问5分会得到同样的前5分:
>>> engine.reset()
>>> engine.random(5)
array([[0.22166437, 0.07980522], # random
[0.72166437, 0.93165708],
[0.47166437, 0.41313856],
[0.97166437, 0.19091633],
[0.01853937, 0.74647189]])
在这里,我们将序列向前推进,以获得相同的第二组5个点:
>>> engine.reset()
>>> engine.fast_forward(5)
>>> engine.random(5)
array([[0.51853937, 0.52424967], # random
[0.26853937, 0.30202745],
[0.76853937, 0.857583 ],
[0.14353937, 0.63536078],
[0.64353937, 0.01807683]])
制作QMC引擎,即子类化 QMCEngine¶
做你自己的 QMCEngine ,必须定义一些方法。以下是一个包装示例 numpy.random.Generator 。
>>> import numpy as np
>>> from scipy.stats import qmc
>>> class RandomEngine(qmc.QMCEngine):
... def __init__(self, d, seed=None):
... super().__init__(d=d, seed=seed)
... self.rng = np.random.default_rng(self.rng_seed)
...
...
... def random(self, n=1):
... self.num_generated += n
... return self.rng.random((n, self.d))
...
...
... def reset(self):
... self.rng = np.random.default_rng(self.rng_seed)
... self.num_generated = 0
... return self
...
...
... def fast_forward(self, n):
... self.random(n)
... return self
然后,我们将其用作任何其他QMC引擎:
>>> engine = RandomEngine(2)
>>> engine.random(5)
array([[0.22733602, 0.31675834], # random
[0.79736546, 0.67625467],
[0.39110955, 0.33281393],
[0.59830875, 0.18673419],
[0.67275604, 0.94180287]])
>>> engine.reset()
>>> engine.random(5)
array([[0.22733602, 0.31675834], # random
[0.79736546, 0.67625467],
[0.39110955, 0.33281393],
[0.59830875, 0.18673419],
[0.67275604, 0.94180287]])