数据导入Galaxy
有三种主要方法可以用数据文件填充您的星系历史,另外还有一种方法可以在星系中共享数据。
从计算机上载文件
文件的数据上传 小于2 GB 你电脑上的这个谎言是相当直截了当的:点击类别“获取数据”并选择工具“上传文件”。然后通过“浏览”按钮选择文件。
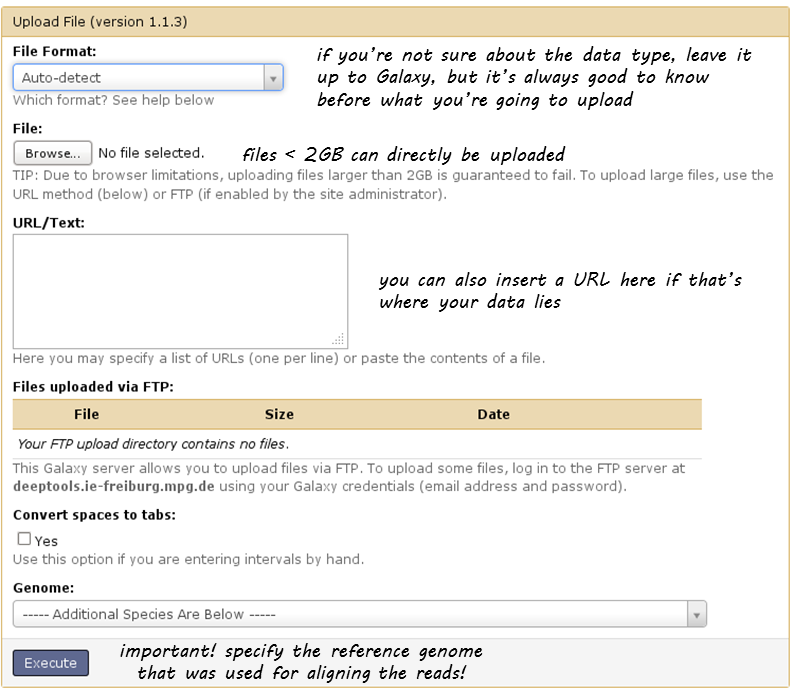
对于文件 大于2GB ,有通过FTP服务器上传的选项。如果您的数据是通过链接到FTP服务器的URL提供的,您可以简单地将该URL粘贴到空文本框中。
如果您没有访问FTP服务器的权限,您可以直接上传到Galaxy的FTP。
在deeptools.ie-freiburg.mpg.de注册(通过“用户”“注册”;注册需要电子邮件地址,免费)
您还需要一个ftp客户机,例如 filezilla .
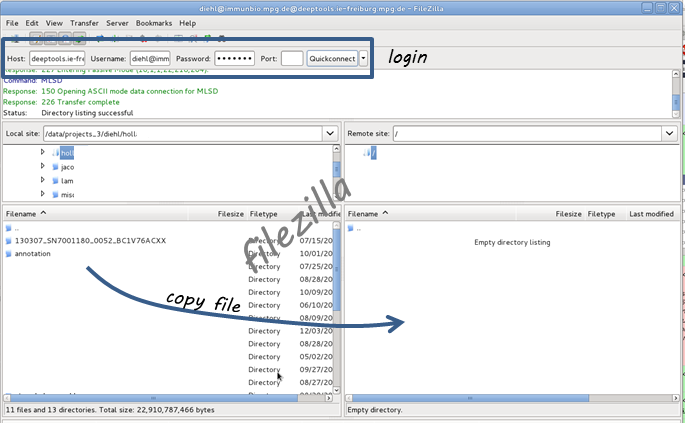
然后登录到 FTP客户端 使用您的 Deeptools Galaxy用户名和密码 (主持人:Deeptools.ie Freiburg.mpg.de)。在下面,你可以看到一个关于FileZilla的屏幕截图。
复制要上载到远程站点的文件(在FileZilla中,只需将文件拖到右侧的窗口即可)
回到 deepTools Galaxy .
点击工具“上传文件”(“通过ftp上传的文件”)-这里,您刚刚通过filezilla复制的文件应该出现。选择所需的文件并点击“执行”。它们将从ftp服务器移到您的历史记录中(即,一旦上传成功,它们将从ftp中删除)。
从Galaxy数据库导入数据集
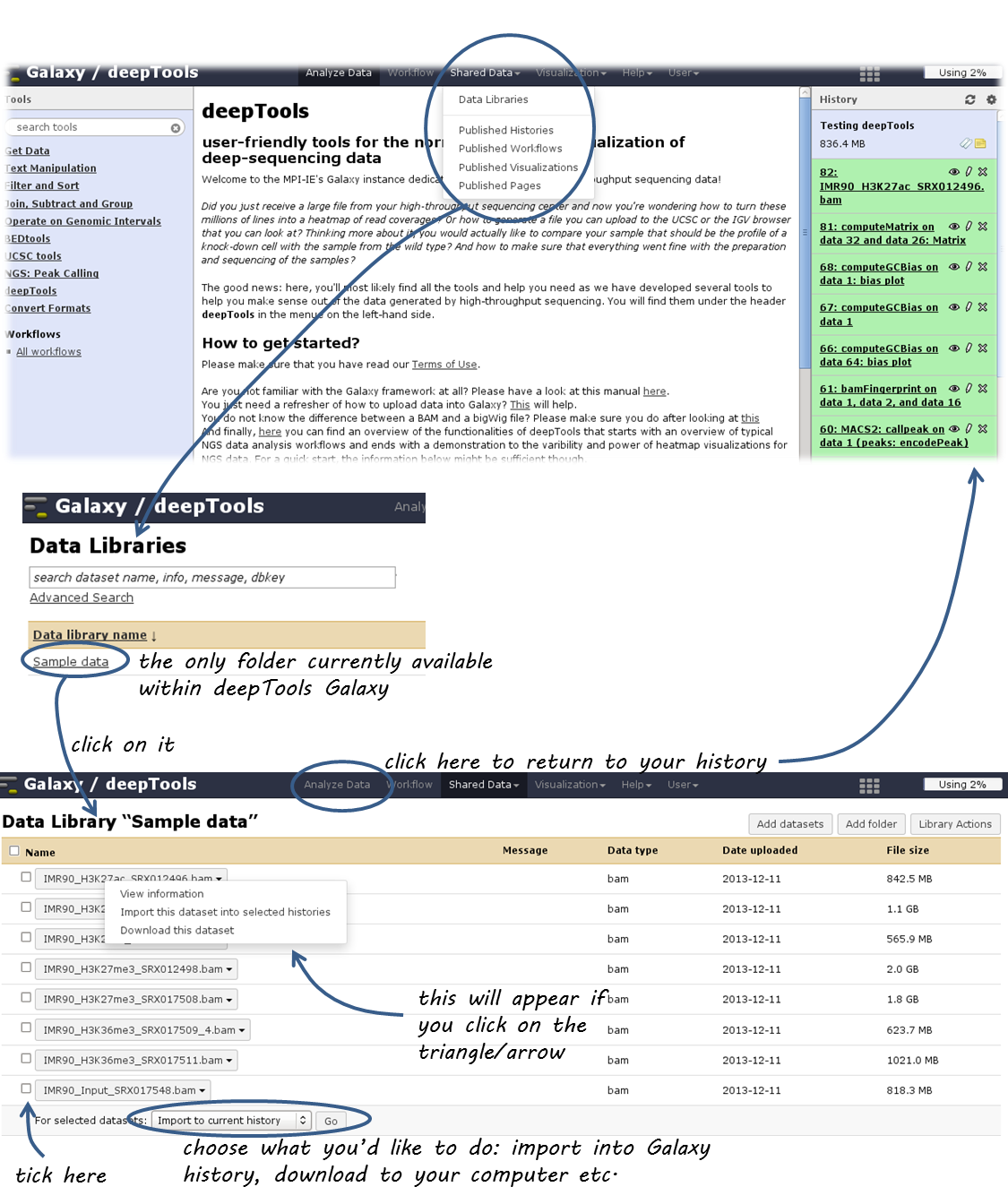
如果您想使用示例数据,可以导入我们保存在Deeptools Galaxy服务器的常规数据存储中的文件。每个人都可以将它们导入到自己的历史记录中,它们不会贡献给用户的磁盘配额。
您可以通过顶部菜单中的“共享数据”访问数据库,然后选择“数据库”。
在数据库中,您将找到一个名为“示例数据”的文件夹,其中包含我们从 Roadmap project 和 UCSC 更准确地说,我们没有 [FASTQ] [] files of various ChIP-seq samples and the corresponding input and mapped the reads to the human reference genome (version hg19) to obtain the [BAM] []您看到的文件。此外,您还可以找到使用 bamCoverage 以及一些BED格式的注释文件以及RNA序列数据。
备注
为了使文件大小保持较小,所有文件只包含19号染色体和X号染色体的数据!
从公共数据库下载注释文件
在许多情况下,你会想查询你的测序数据结果以获得已知的基因组注释,例如基因、外显子、转录起始位点等。这些信息可以通过基因组注释的两个主要来源获得。 UCSC 和 BioMart .
警告
UCSC和Biomart适应不同的基因组注释方式,即UCSC中定义的基因可能与从Biomart下载的基因文件中的相同区域不对应。(对于基因组注释问题的简要概述,您可以查看 Wikipedia 如果你一直想更多地了解这些问题, this 可能是个好的开始。)
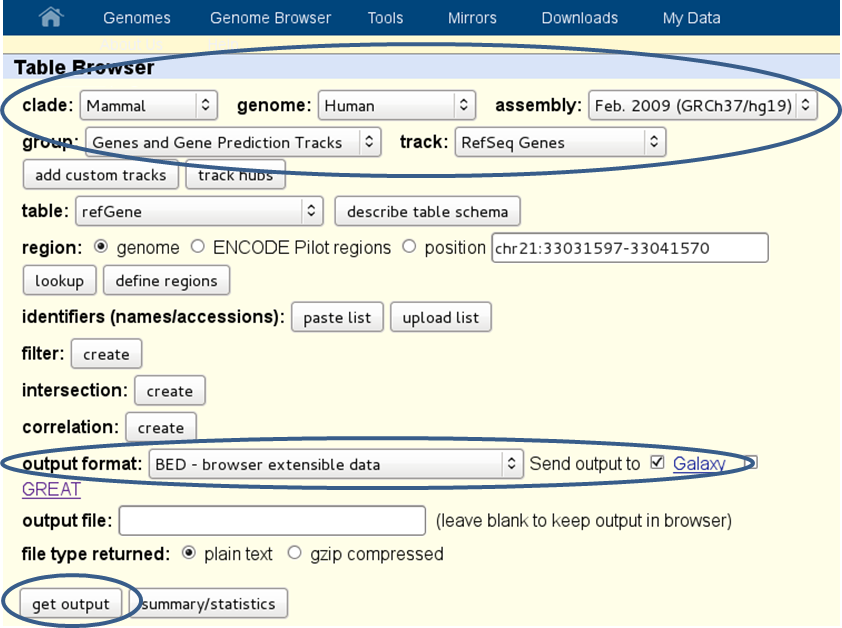
您可以通过我们的Galaxy实例方便地访问存储在UCSC或Biomart上的数据,该实例将生成的文件导入到您的历史记录中。去吧。 “获取数据” “UCSC”或“Biomart”。
大多数注释文件可能位于 [BED] []格式,但是,您也可以找到其他数据集。例如,UCSC提供了一系列数据,您可以通过“组”和“跟踪”菜单浏览这些数据(例如,您可以通过“组”菜单(“映射和排序跟踪”)从UCSC下载基因组的GC内容作为信号文件。
警告
通过这个接口下载的每个文件限制在100000行,这可能不足以满足某些哺乳动物的数据集。
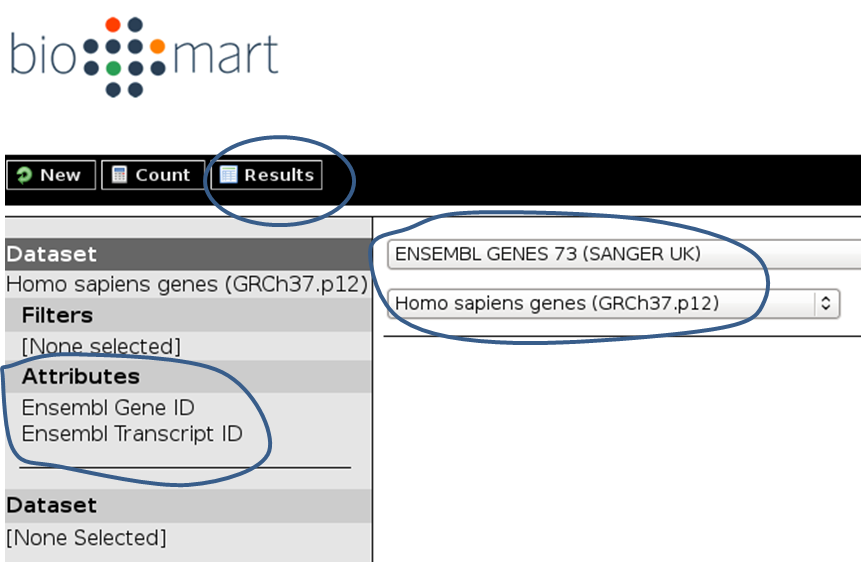
以下是下载为人类基因组定义的所有refseq基因(hg19版本)的bed文件的截图:
以下是您在生物艺术方法中的做法:
小技巧
默认情况下, Biomart不会输出床位文件 就像加州大学旧金山分校那样。因此,确保通过“属性”部分获得所需的所有信息(很可能是:染色体、基因开始、基因结束、ID、链)非常重要。您可以随时单击“结果”按钮,检查将发送到Galaxy的表的格式(请注意,链信息将被解码为1表示“前进”或“加”链,而-1表示“后退”或“减”链)。
警告
请注意,UCSC中的床位文件将带有标记为“chr”的染色体,而ENSEMBL通常只返回数字-这可能会导致不兼容,即在处理UCSC和ENSEMBL中的注释时,需要确保使用相同的命名!
在历史记录之间复制数据集
如果你注册了Deeptools Galaxy,你可以有多个历史。
为了尽量减少您占用的磁盘空间,我们强烈建议 copy 当您在不同历史中使用相同的数据集时,历史之间的数据集。
备注
只有注册用户才能复制数据集。
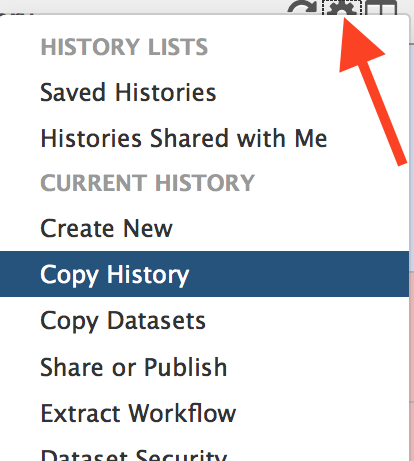
复制可以通过历史面板轻松完成 option 按钮“复制数据集”。在主框架中,您现在应该能够选择要从左侧复制的历史记录和右侧复制的目标历史记录。
更多帮助
提示
如果遇到故障数据集(用红色标记),请 发送错误报告 通过Galaxy Bug报告按钮,如果您指出您的电子邮件地址,我们将与您联系。
一般Galaxy使用帮助 |
|
我们的特定Galaxy实例经常遇到的问题 |
|
对于常见问题解答中未解决的问题 |
deepTools Galaxy <http://deeptools.ie-freiburg.mpg.de> _. |
code @ github <https://github.com/deeptools/deepTools/> _. |