Deeptools地块画廊
我们正试图收集使用deeptools生成的各种绘图。对于我们自己创建的情节,我们试图指出用于创建每个图像的选项,所以也许这些可以作为您的灵感。
归一化芯片序列信号和峰值区域
此图像由发布 Ibrahim et al., 2014 (NAR) . 他们使用deeptools以10个基址的分辨率生成每千基址每百万次读取的扩展读取,并在 IGV .
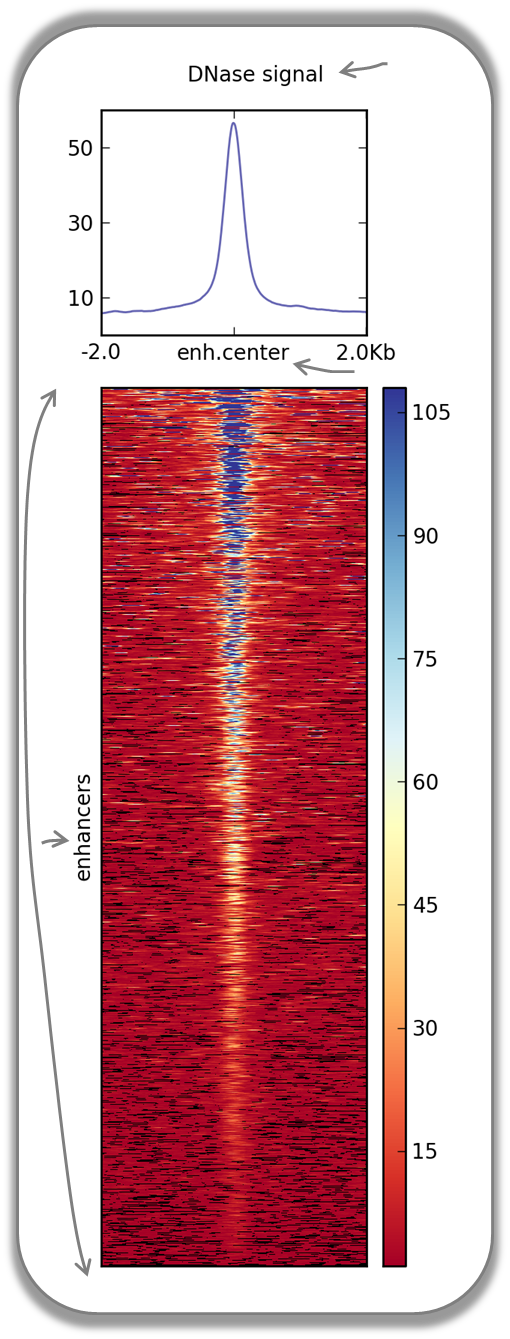
小鼠ES细胞中增强子的dnase可及性
下图显示了增强子区域通常是高度易接近的染色质的小片段(例如,可以找到增强子的更多信息, here )在heatmap中,黄色和蓝色的图块表示大量被排序的读取(表示开放染色质),而黑点表示缺少数据点。忽略了Y轴的适当标记。
快速事实:
computeMatrix 模式:参考点
区域文件 :具有典型增强器区域的bed文件 Whyte et al., 2013 (下载 here )
信号文件 :bigwig文件 DNase signal from UCSC
热疗化妆品 :标签、标题、热图高度
命令:
$ computeMatrix reference-point \
-S DNase_mouse.bigwig \
-R Whyte_TypicalEnhancers_ESC.bed \
--referencePoint center \
-a 2000 -b 2000 \ ## regions before and after the enhancer centers
-out matrix_Enhancers_DNase_ESC.tab.gz
$ plotHeatmap \
-m matrix_Enhancers_DNase_ESC.tab.gz\
-out hm_DNase_ESC.png \
--heatmapHeight 15 \
--refPointLabel enh.center \
--regionsLabel enhancers \
--plotTitle 'DNase signal' \
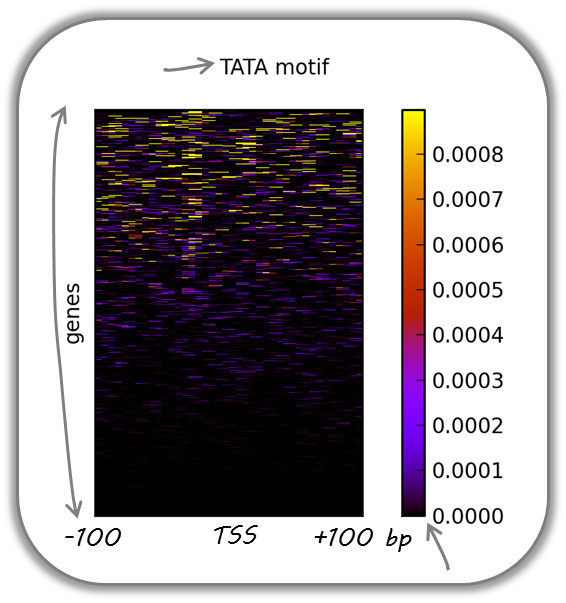
塔塔盒丰富了小鼠基因的TSS。
使用 TRAP 套房,我们产生了一个大的文件,其中包含了陷阱分数为众所周知的塔塔盒基序沿小鼠基因组。trap评分是对给定DNA序列中蛋白质-DNA相互作用强度的测量;评分越高,基序越接近一致基序序列。以下热图表明:
塔塔式图案经常出现
在许多小鼠基因的TSS的稍上游有明显的TATA基序聚集。
有许多基因在其启动子处不包含塔塔样的基序。
注意,热图显示 all 小鼠refseq基因,所以大约15000个基因!
快速事实:
computeMatrix mode :参考点
区域文件 :包含所有鼠标基因的床文件(来自UCSC表格浏览器)
信号文件 :Tata PSEM分数的Bigwig文件
热疗化妆品 :配色方案、标签、标题、热图高度,仅显示热图+颜色条
命令:
$ computeMatrix reference-point \
-S TATA_01_pssm.bw \
-R RefSeq_genes.bed \
--referencePoint TSS \
-a 100 -b 100 \
--binSize 5 \
$ plotHeatmap \
-m matrix_Genes_TATA.tab.gz \
-out hm_allGenes_TATA.png \
--colorMap hot_r \
--missingDataColor .4 \
--heatmapHeight 7 \
--plotTitle 'TATA motif' \
--whatToShow 'heatmap and colorbar' \
--sortRegions ascend
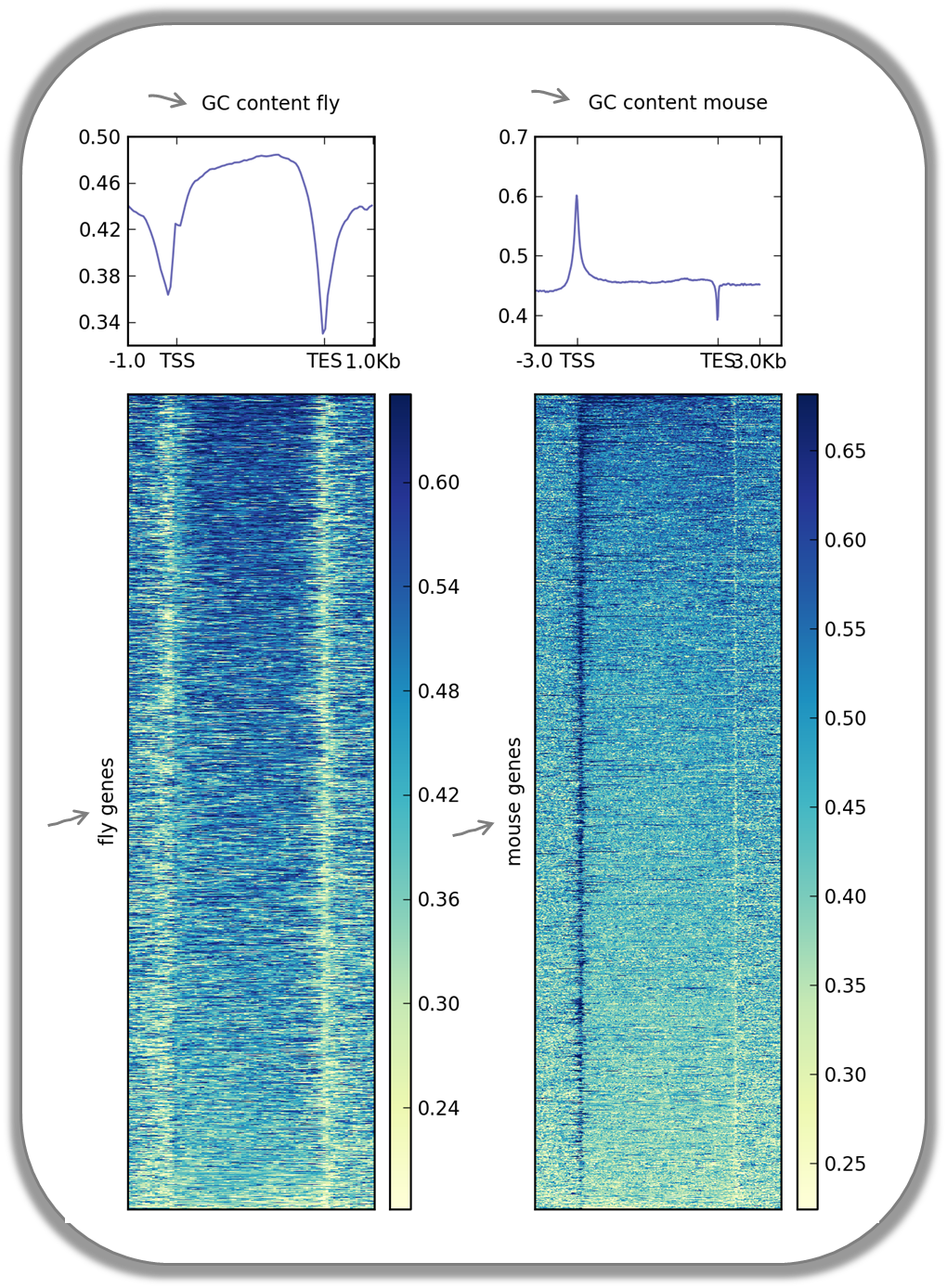
小鼠和蝇基因GC含量的可视化
众所周知,不同物种的基因组GC含量不同。在这里,我们使用了两个bigwig文件,计算了小鼠和果蝇基因组中50个碱基窗口的GC含量,并对基因区域显示了结果分数。
这些图片很好地说明了苍蝇和老鼠中完全相反的GC分布:虽然哺乳动物基因组的基因开始富含GS和CS,但苍蝇启动子显示GC含量的减少。
快速的事实 |
|
|---|---|
ComputeMatrix模式 |
缩放区域 |
区域文件 |
带有鼠标和飞行基因的BED文件(来自UCSC表格浏览器) |
信号文件 |
包含gc内容的bigwig文件 |
热疗化妆品 |
颜色方案、标签、标题、丢失数据的颜色设置为白色、热图高度。 |
由于这两个物种基因的中位数不同(基因 D.melanogaster 内含子较少,比哺乳动物基因短得多。因此,computematrix必须使用稍微不同的参数运行,而plothotmap命令实际上是相同的(除了标签)。
$ computeMatrix scale-regions \
-S GCcontent_Mm9_50_5.bw \
-R RefSeq_genes_uniqNM.bed \
-bs 50
-m 10000 -b 3000 -a 3000 \
-out matrix_GCcont_Mm9_scaledGenes.tab.gz \
--skipZeros \
--missingDataAsZero
$ computeMatrix scale-regions \
-S GCcontent_Dm3_50_5.bw \
-R Dm530.genes.bed \
-bs 50
-m 3000 -b 1000 -a 1000 \
-out matrix_GCcont_Dm3_scaledGenes.tab.gz \
--skipZeros --missingDataAsZero
$ plotHeatmap \
-m matrix_GCcont_Dm3_scaledGenes.tab.gz \
-out hm_GCcont_Dm3_scaledGenes.png \
--colorMap YlGnBu \
--regionsLabel 'fly genes' \
--heatmapHeight 15 \
--plotTitle 'GC content fly' &
$ plotHeatmap \
-m matrix_GCcont_Mm9_scaledGenes.tab.gz \
-out hm_GCcont_Mm9_scaledGenes.png \
--colorMap YlGnBu \
--regionsLabel 'mouse genes' \
--heatmapHeight 15 \
--plotTitle 'GC content mouse' &
两种不同细胞类型小鼠转录起始点周围的CpG甲基化
除了组蛋白尾的甲基化,胞嘧啶也可以甲基化(有关CpG甲基化的更多信息,请阅读 here )在哺乳动物基因组中,大多数CpG是甲基化的,除非它们在基因启动子中,需要保持非甲基化以允许完整的转录活性。在下面的热图中,我们使用主要在ES细胞中表达的基因,并检查转录起始位点周围甲基化胞嘧啶的百分比。蓝色信号表明甲基化胞嘧啶很少被发现。当你比较ES细胞和神经祖细胞(NP)之间的CpG甲基化信号时,你可以看到大多数基因仍然是非甲基化的,但是总的来说,TSS周围的CpG甲基化量增加,这是由更强的红色信号和Cp的轻微升高所指示的。G甲基化信号在总结图中。这支持了一种观点,即存储在bed文件中的基因确实比在np细胞中更倾向于在es中表达。
这张照片是从 Chelmicki & Dündar et al. (2014), eLife .
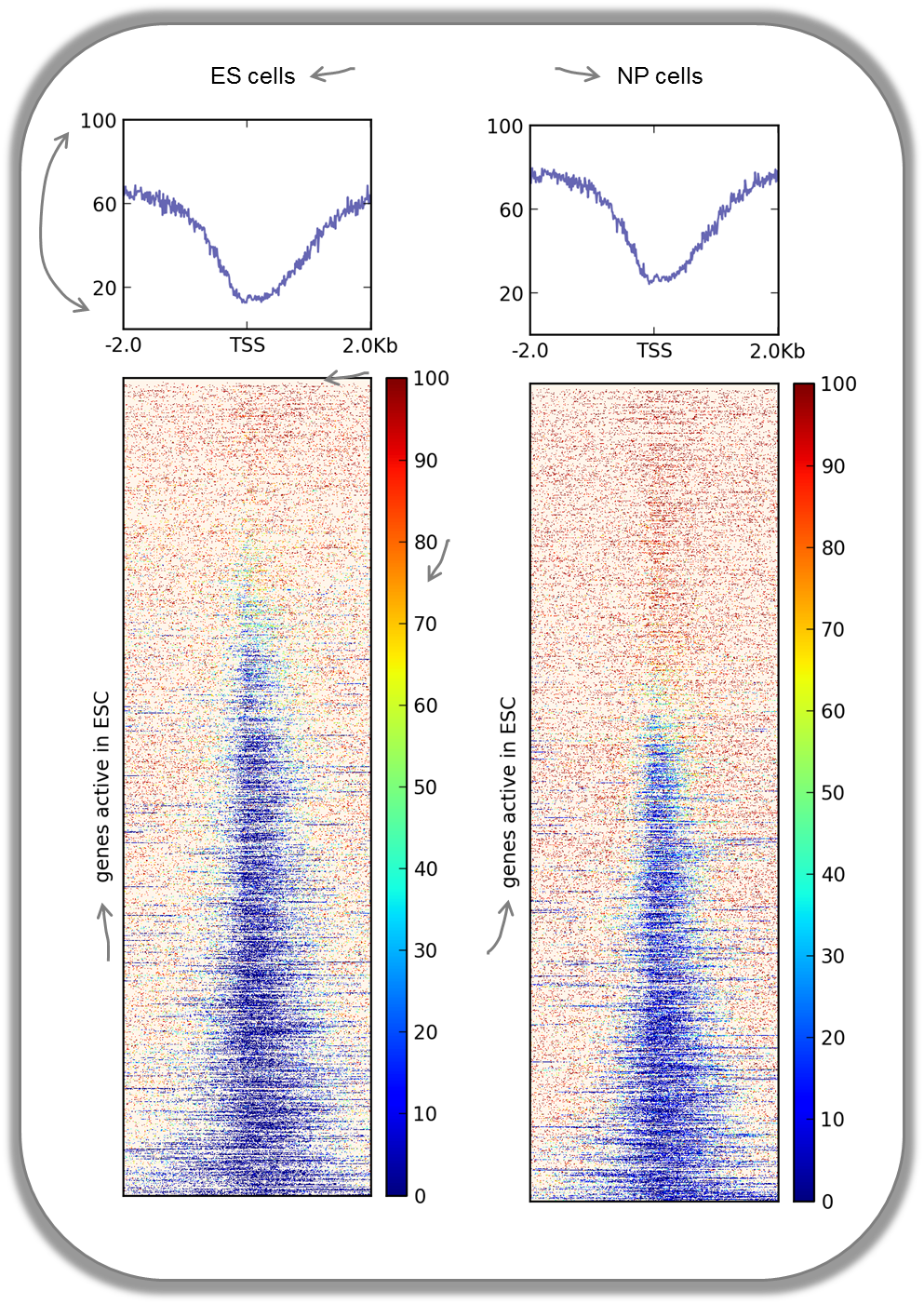
快速的事实 |
|
|---|---|
ComputeMatrix模式 |
参考点 |
区域文件 |
BED ES细胞中表达的文件鼠基因 |
信号文件 |
bigWig 含有甲基化胞嘧啶部分的文件(来自 Stadler et al., 2011 ) |
热疗化妆品 |
颜色方案、标签、标题、丢失数据的颜色设置为自定义颜色、纵断面的Y轴更改、热图高度 |
ES和NP单元中的bigwig文件命令相同:
$ computeMatrix reference-point \
-S GSE30202_ES_CpGmeth.bw \
-R activeGenes_ESConly.bed \
--referencePoint TSS \
-a 2000 -b 2000 \
-out matrix_Genes_ES_CpGmeth.tab.gz
$ plotHeatmap \
-m matrix_Genes_ES_CpGmeth.tab.gz \
-out hm_activeESCGenes_CpG_ES_indSort.png \
--colorMap jet \
--missingDataColor "#FFF6EB" \
--heatmapHeight 15 \
--yMin 0 --yMax 100 \
--plotTitle 'ES cells' \
--regionsLabel 'genes active in ESC'
蚊子基因组蛋白标记 冈比亚按蚊
这个数字是从 Gómez-Díaz et al. (2014): Insights into the epigenomic landscape of the human malaria vector *Anopheles gambiae*. From Genet Aug15;5:277 . 它显示了h3k27me3(左)和h3k27ac(右)在基因特征上的分布。 冈比亚按蚊 中肠。相对于染色质输入显示富集或耗尽。图中的区域包括基因体,在TSSS和TTSS的5'端有200个碱基片段。上面显示了每个组蛋白修饰的基因区域的平均分布±200个碱基。

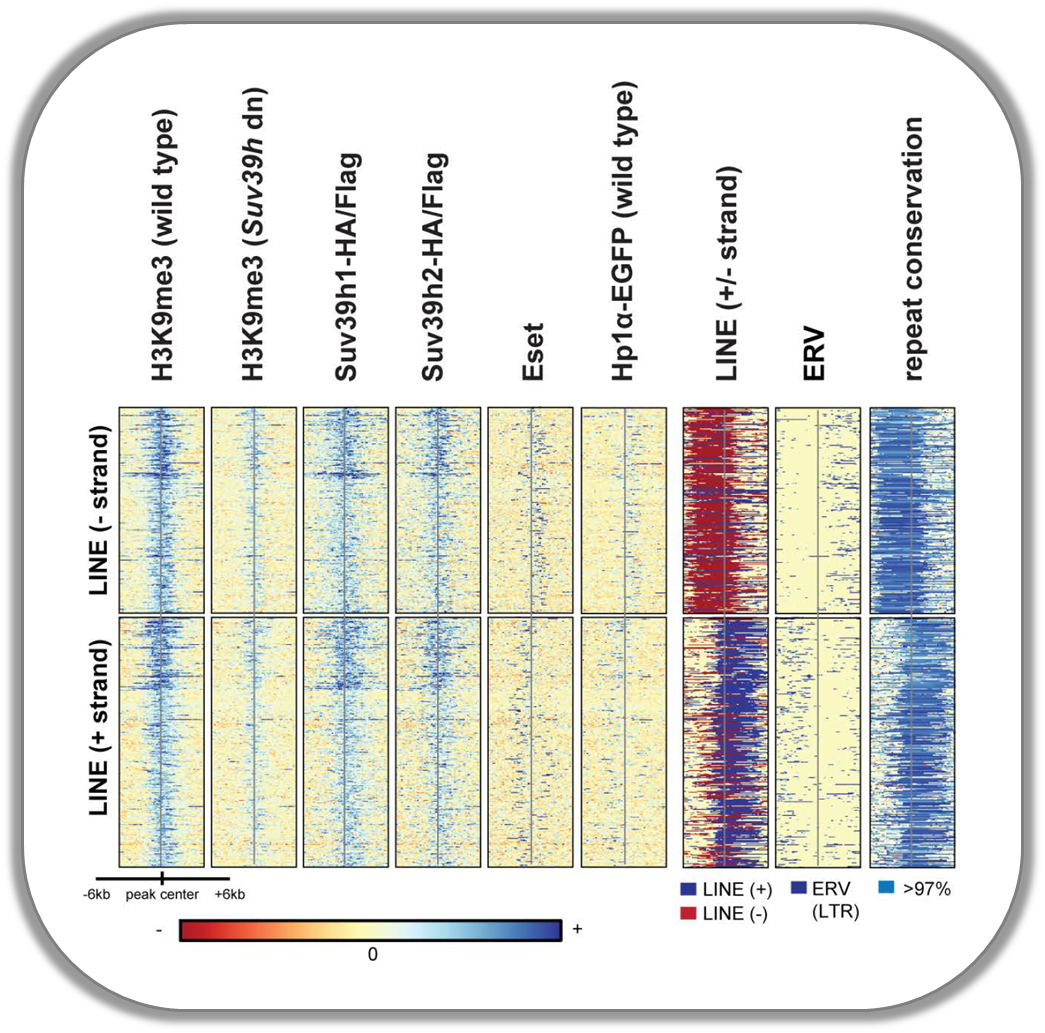
抑制染色质标记及其酶和重复元素保存分数的信号
这张图片来自 Bulut-Karsliogu and De La Rosa-Velázquez et al. (2014), Mol Cell. heatmap描述了与抑制染色质相关的蛋白和组蛋白标记的未标记峰区域的各种信号类型。将峰分为正反向链上的长分散元素(线)。这些信号包括h3k9me3、suv39h1、suv39h2、eset和hp1alpha egfp的标准化chip seq信号,然后是line和erv内容和重复保存分数。
deepTools Galaxy <http://deeptools.ie-freiburg.mpg.de> _. |
code @ github <https://github.com/deeptools/deepTools/> _. |