带标记的图像函数¶
带标签的图像是整数图像,其中的值对应于不同的区域。也就是说,区域1是所有有值的像素 1 区域2是值为2的像素,依此类推。按照惯例, region 0 is the background and often handled differently 。
为图像添加标签¶
在 0.6.5 版本加入.
第一步是从二元函数中获得标记函数:
import mahotas as mh
import numpy as np
from pylab import imshow, show
regions = np.zeros((8,8), bool)
regions[:3,:3] = 1
regions[6:,6:] = 1
labeled, nr_objects = mh.label(regions)
imshow(labeled, interpolation='nearest')
show()
(Source code, png, hires.png, pdf)
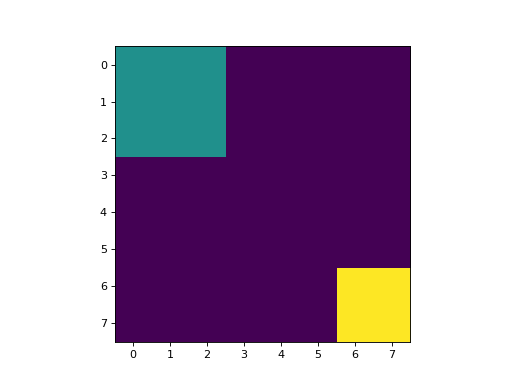
这将产生一个具有3个值的图像:
背景,其中原始图像为0
第一个区域:(0:3,0:3);
第二个地区:(6:,6:)。
还有一个额外的论据 label :结构元素,默认为3x3交叉(或4邻域)。这定义了两个像素位于同一区域意味着什么。您可以使用8个邻域,将其替换为一个正方形::
labeled,nr_objects = mh.label(regions, np.ones((3,3), bool))
我们现在可以收集有关已标记区域的一些统计数据。例如,它们有多大?
sizes = mh.labeled.labeled_size(labeled)
print('Background size', sizes[0])
print('Size of first region: {}'.format(sizes[1]))
此大小简单地用每个区域中的像素数来衡量。相反,我们可以测量每个区域的总权重:
array = np.random.random_sample(regions.shape)
sums = mh.labeled_sum(array, labeled)
print('Sum of first region: {}'.format(sums[1]))
滤波区¶
在 0.9.6 版本加入: remove_regions & relabel 都被添加了。
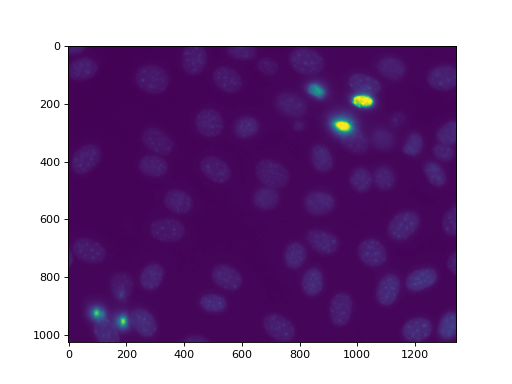
这里有一个稍微复杂一点的例子。完整的代码在 demos directory AS nuclear.py. We are going to use this image, a fluorescent microscopy image from a nuclear segmentation benchmark
此图像以以下形式提供 mahotas.demos.nuclear_image()
import mahotas as mh
import mahotas.demos
import numpy as np
from pylab import imshow, show
f = mh.demos.nuclear_image()
f = f[:,:,0]
imshow(f)
show()
(Source code, png, hires.png, pdf)
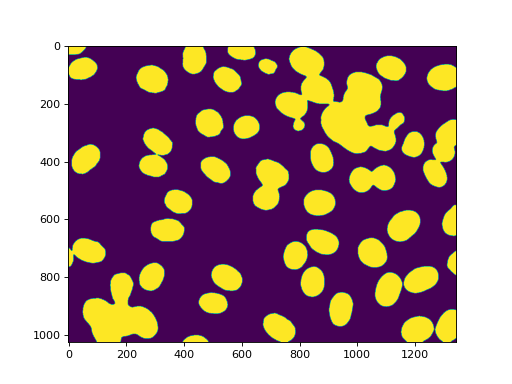
首先,我们执行一些高斯滤波和阈值处理:
f = mh.gaussian_filter(f, 4)
f = (f> f.mean())
(如果不使用高斯过滤器,则得到的阈值图像具有非常嘈杂的边缘。您可以在 demos/ 目录,并试用它。)
f = mh.gaussian_filter(f, 4)
f = (f> f.mean())
imshow(f)
show()
(Source code, png, hires.png, pdf)
标记得到了我们所有的核::
labeled, n_nucleus = mh.label(f)
print('Found {} nuclei.'.format(n_nucleus))
labeled, n_nucleus = mh.label(f)
print('Found {} nuclei.'.format(n_nucleus))
imshow(labeled)
show()
(Source code, png, hires.png, pdf)
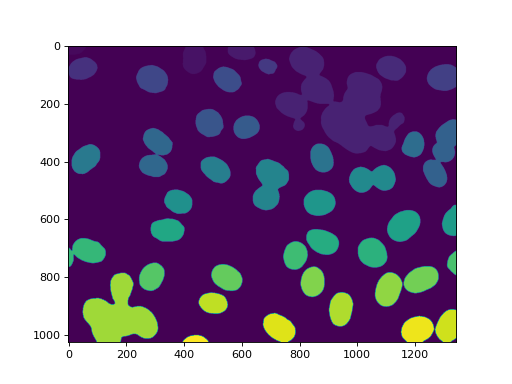
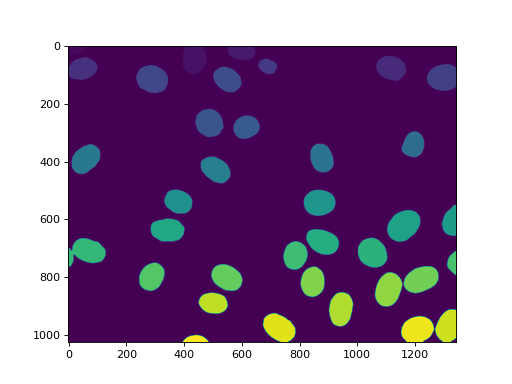
42 发现了细胞核。没有遗漏,但不幸的是,我们也得到了一些聚合。在这种情况下,我们将假设我们想要对真实的原子核进行一些测量,但愿意过滤掉任何不是完整的原子核或原子核上的肿块。因此,我们测量大小并过滤:
sizes = mh.labeled.labeled_size(labeled)
too_big = np.where(sizes > 10000)
labeled = mh.labeled.remove_regions(labeled, too_big)
sizes = mh.labeled.labeled_size(labeled)
too_big = np.where(sizes > 10000)
labeled = mh.labeled.remove_regions(labeled, too_big)
imshow(labeled)
show()
(Source code, png, hires.png, pdf)
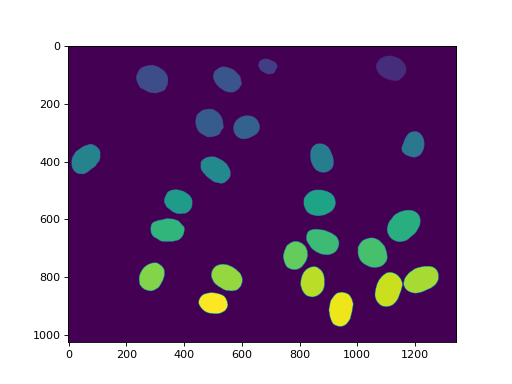
我们还可以删除与边界接触的区域::
labeled = mh.labeled.remove_bordering(labeled)
labeled = mh.labeled.remove_bordering(labeled)
imshow(labeled)
show()
(Source code, png, hires.png, pdf)
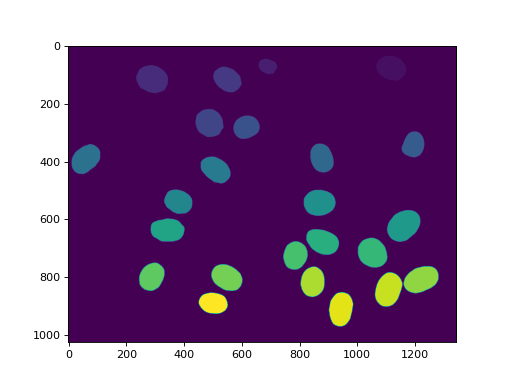
这个数组, labeled 现在具有范围内的值 0 至 n_nucleus ,但缺少某些值(例如,If Region 7 是接触边境的人之一,然后 7 未在标签中使用)。我们可以的 relabel 要获得更干净的版本::
relabeled, n_left = mh.labeled.relabel(labeled)
print('After filtering and relabeling, there are {} nuclei left.'.format(n_left))
现在,我们有 24 原子核和 relabeled 从 0 (背景)至 24 。
relabeled, n_left = mh.labeled.relabel(labeled)
print('After filtering and relabeling, there are {} nuclei left.'.format(n_left))
imshow(relabeled)
show()
(Source code, png, hires.png, pdf)
在Mahotas之后的版本中 1.4 ,我们甚至可以通过单个调用来执行许多相同的操作 mh.labeled.filter_labeled **
relabeled,n_left = mh.labeled.filter_labeled(labeled, remove_bordering=True, max_size=10000)
边框¶
边界像素是指在其邻近区域中有多个区域的像素(其中一个区域可以是背景)。
方法检索边界像素。 borders() 函数,该函数获取所有边框或 border() (请注意单数),它只获取一对区域之间的边界。与往常一样,邻居的意思是由结构元素定义的,默认为3x3十字。
API文档¶
这个 mahotas.labeled 子模块包含上述功能。 label() 也可以通过以下方式获得 mahotas.label 。
- mahotas.labeled.bbox(f, as_slice=False)
带标签的数组中所有对象的边界框。
之后::
bboxes = mh.labeled.bbox(f)
bboxes[34]将包含(f == 34)。- 参数:
- f整数ndarray
- as_slice布尔型,可选
是否返回切片对象而不是整数坐标(默认值:FALSE)。
- 退货:
- bboxesNdarray
参见
mh.bbox此函数的二进制版本
- mahotas.labeled.border(labeled, i, j, Bc={3x3 cross}, out={np.zeros(labeled.shape, bool)}, always_return=True)
计算之间的边界区域 i 和 j 地区。
如果像素有值,则它位于边框上 i (或 j )和其附近的像素(由 Bc )有价值 j (或 i )。
- 参数:
- labeled整数类型的ndarray
输入标记数组
- i整数
- j整数
- Bc结构元素,可选
- out :ndarray形状相同 labeled ,dtype=bool,可选与形状相同的ndarray
存储输出的位置。如果
None,则分配一个新的数组- always_return布尔值,可选
如果为False,则在边界上没有像素的情况下,返回
None。否则(默认设置),它始终返回一个数组,即使该数组为空。
- 退货:
- border_img布尔ndarray
像素是True,恰好是在 i 和 j 在……里面 labeled
- mahotas.labeled.borders(labeled, Bc={3x3 cross}, out={np.zeros(labeled.shape, bool)})
计算边框像素
如果像素有值,则它位于边框上 i 以及其邻域中的像素(由 Bc )有价值 j 同
i != j。- 参数:
- labeled整数类型的ndarray
输入标记数组
- Bc结构元素,可选
- out :ndarray形状相同 labeled ,dtype=bool,可选与形状相同的ndarray
存储输出的位置。如果
None,则分配一个新的数组- mode{'reflect', 'nearest', 'wrap', 'mirror', 'constant' [default], 'ignore'}
如何处理边框
- 退货:
- border_img布尔ndarray
像素在有边框的地方为True labeled
- mahotas.labeled.bwperim(bw, n=4)
找出二值图像中对象的周长。
如果像素的值为1,并且其邻域中至少有一个零值像素,则该像素是对象周长的一部分。
默认情况下,像素的邻域是最近的4个像素,但如果 n 设置为8,则将考虑最近的8个像素。
- 参数:
- bwNdarray
黑白图像(任何其他图像都将转换为黑白图像)
- n整型,可选
连通性。必须是4或8(默认值:4)
- mode{'reflect', 'nearest', 'wrap', 'mirror', 'constant' [default], 'ignore'}
如何处理边框
- 退货:
- perimNdarray
布尔型图像
参见
borders函数这是一个更一般的函数
- mahotas.labeled.filter_labeled(labeled, remove_bordering=False, min_size=None, max_size=None)
基于一系列条件过滤标注区域
在 1.4.1 版本加入.
- 参数:
- labeled标号数组
- remove_bordering布尔值,可选
是否删除接触边界的区域
- min_size整型,可选
要保留的对象的最小大小(以像素为单位)(默认为无最小值)
- max_size整型,可选
要保留的对象的最大大小(以像素为单位)(默认为无最大值)
- 退货:
- filtered标号数组
- nr集成
新标签数量
- mahotas.labeled.is_same_labeling(labeled0, labeled1)
检查是否
labeled0和labeled1表示相同的标签(即,除了标签值可能发生变化外,它们是否相同)。请注意,背景(值0)的处理方式不同。即
IS_SAME_LABELING(a,b)表示np.all((a==0)==(b==0))
- 参数:
- labeled0Ndarray of int
带标签的数组
- labeled1Ndarray of int
带标签的数组
- 退货:
- same布尔尔
如果作为参数传递的标注相等,则为True
参见
label功能
relabel功能
- mahotas.labeled.label(array, Bc={3x3 cross}, output={new array})
标记数组,它被解释为二进制数组
这也被称为 connected component labeled ,其中连接性由结构化元素定义
Bc。请参阅:https://en.wikipedia.org/wiki/Connected-component_labeling
- 参数:
- arrayNdarray
这将被解释为二进制数组
- BcNdarray,可选
这是要使用的结构化元素
- outNdarray,可选
输出数组。必须是np.int32类型的C数组
- 退货:
- labeledNdarray
标记的结果
- nr_objects集成
对象数量
- mahotas.labeled.labeled_max(array, labeled, minlength=None)
标记为最小。
mins将是一个大小为labeled.max() + 1,在哪里mins[i]等于np.min(array[labeled == i])。- 参数:
- array任何类型的ndarray
- labeled集成ndarray
标签地图。这与从返回的类型相同
mahotas.label()
- 退货:
- mins :一维ndarray,共
array.dtype一维阵列,共
- mins :一维ndarray,共
- mahotas.labeled.labeled_size(labeled)
等同于:
for i in range(...): sizes[i] = np.sum(labeled == i)
但是,自然地,速度要快得多。
- 参数:
- labeled集成ndarray
- 退货:
- sizes一维整数线阵
参见
mahotas.fullhistogram函数使用另一个名称几乎相同的函数(唯一的区别是该函数只接受无符号整数类型)。
- mahotas.labeled.labeled_sum(array, labeled, minlength=None)
标记为SUM。Sum将是一个大小为
labeled.max() + 1,在哪里sum[i]等于np.sum(array[labeled == i])。- 参数:
- array任何类型的ndarray
- labeled集成ndarray
标签地图。这与从返回的类型相同
mahotas.label()- minlength整型,可选
返回数组的最小大小。如果标签的数量少于
minlength区域,0被添加到结果中。(可选)
- 退货:
- sums :一维ndarray,共
array.dtype一维阵列,共
- sums :一维ndarray,共
- mahotas.labeled.perimeter(bwimage, n=4, mode='constant')
计算二值图像中所有对象的总周长。
- 参数:
- bwimage数组
二值图像
- n整型,可选
已传递给
bwperim原样- mode字符串,可选
已传递给
bwperim原样
- 退货:
- p浮动
二值图像中所有对象的总周长
参见
bwperim函数查找周长区域
参考文献
[1]本克里德,D.克鲁克斯。周长估计器的设计与FPGA实现。贝尔法斯特女王大学。Https://www.cs.qub.ac.uk/~d.crookes/webpubs/papers/perimeter.doc
- mahotas.labeled.relabel(labeled, inplace=False)
重新标记可确保
relabeled是一种带标签的图像,因此从1到relabeled.max()被使用(0保留为背景并被传递)。示例::
labeled,n = label(some_binary_map) for region in range(n): if not good_region(labeled, region + 1): # This deletes the region: labeled[labeled == (region + 1)] = 0 relabel(labeled, inplace=True)
- 参数:
- relabeledNdarray of int
带标签的数组
- inplace布尔型,可选
是否就地执行重新标记,擦除中的值
labeled(默认:FALSE)
- 退货:
- 重新标记:ndarray
- nr_objs集成
对象数量
参见
label功能
- mahotas.labeled.remove_bordering(labeled, rsize=1, out={np.empty_like(im)})
删除正在接触边框的对象。
经过
labeledASout实现就地作业。- 参数:
- labeledNdarray
标号数组
- rsize整型或元组,可选
允许对象存活的到边界的最小距离(以曼哈坦距离表示)。可以是整数或元组,其中len==labeled.ndim。
- outNdarray,可选
如果
im被作为out,然后它以内联方式运行。
- 退货:
- slabeledNdarray
的子集
labeled
- mahotas.labeled.remove_regions(labeled, regions, inplace=False)
REMOVERED=REMOVE_REGIONS(已标记,REGIONS,INPLACE=FALSE):
删除中的区域
regions。如果以元素为基础in运算符存在,则等同于::labeled[ labeled element-wise-in regions ] = 0
此函数 does not 重新标记它的论点。您可以使用
relabel函数::removed = relabel(remove_regions(labeled, regions))
或者,保存一个映像分配::
removed = relabel(remove_regions(labeled, regions), inplace=True)
这是相同的,但在重新标记操作中重复使用内存。
- 参数:
- relabeledNdarray of int
带标签的数组
- regions整型序列
这些区域将被移除
- inplace布尔型,可选
是否就地执行删除,擦除中的值
labeled(默认:FALSE)
- 退货:
- removedNdarray
参见
relabel功能删除不必要的区域后,重新标记您的标签图像通常是一个好主意。
- mahotas.labeled.remove_regions_where(labeled, conditions, inplace=False)
基于布尔数组移除区域
如果出现以下情况,则删除区域
conditions[region-id]计算结果为True。此函数 does not 重新标记它的论点。您可以使用
relabel函数::removed = relabel(remove_regions_where(labeled, conditions))
或者,保存一个映像分配::
removed = relabel(remove_regions(labeled, conditions), inplace=True)
这是相同的,但在重新标记操作中重复使用内存。
参见
remove_regions使用整数索引的此函数的函数变体