skbio.stats.ordination.OrdinationResults.plot¶
- OrdinationResults.plot(df=None, column=None, axes=(0, 1, 2), axis_labels=None, title='', cmap=None, s=20)[源代码]¶
创建由元数据着色的排序结果的三维散点图。
状态:从0.4.0开始实验。
创建排序结果的三维散点图,其中每个点代表一个样本。或者,可以通过元数据对这些点进行着色(请参见 df 和 column (见下文)。
- 参数:
df (pd.DataFrame, optional) --
DataFrame包含示例元数据的。必须按样本ID索引,排序结果中的所有样本ID必须存在于DataFrame。如果None,样本(即,点)不会被元数据着色。column (str, optional) -- 中的列名 df 要对样本(即,绘图中的点)进行着色,请使用。不能具有丢失的数据(即,
np.nan)。 column 可以是数字的也可以是绝对的。如果为数字,则该列中的所有值都将强制转换为float并映射到颜色,使用 cmap 。将包括一个颜色条作为图例。如果是绝对的(即,不是 column 可能会被塑造成float),将使用沿线等间距的点为每个类别选择颜色 cmap 。将包括一个传奇故事。如果None,样本(即,点)不会被元数据着色。axes (iterable of int, optional) -- 要在x轴、y轴和z轴上打印的样本坐标的索引。例如,如果绘制PCoA结果,
axes=(0, 1, 2)将在x轴上绘制PC 1,在y轴上绘制PC 2,在z轴上绘制PC 3。必须恰好包含三个元素。axis_labels (iterable of str, optional) -- X轴、y轴和z轴的标签。如果
None,标签将是的值 axes 演绎成弦乐。title (str, optional) -- 剧情标题。
cmap (str or matplotlib.colors.Colormap, optional) -- 要用于贴图的matplotlib色彩贴图的名称或实例 column 值设置为颜色。如果
None,默认为matplotlib RC文件中指定的颜色映射。定性色图(例如,Set1)被推荐用于分类数据,而顺序色彩映射(例如,Greys),建议用于数字数据。看见 [1] 用于这些色彩映射表分类。s (scalar or iterable of scalars, optional) -- 点的大小。请参阅matplotlib的
Axes3D.scatter文档以了解更多详细信息。
- 返回:
该图包含散点图和图例/颜色条(如果提供了元数据)。
- 返回类型:
- 抛出:
ValueError -- 在无效输入时引发,包括以下情况:-没有至少三个要绘制的维度-没有恰好有三个值 axes 、它们不是唯一的,或者超出范围-- axis_labels -任一 df 或 column 是在没有另一个的情况下提供的- column 不在
DataFrame-排序结果中的样本ID不在 df 或中有缺失的数据 column
参见
mpl_toolkits.mplot3d.Axes3D.scatter备注
该方法创建排序结果的基本曲线图,并且旨在提供对元数据上下文中的结果的快速查看(例如,从IPythonNotebook内)。为了更好地进行定制,并生成出版质量的数据,我们推荐Emperior [2].
引用
示例
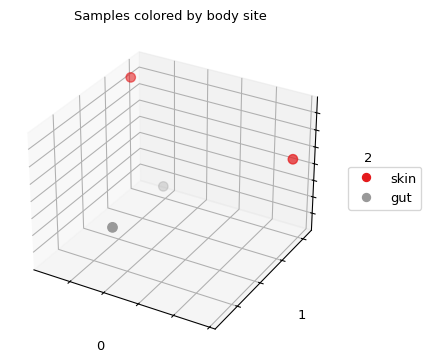
定义一个包含四个标记为A-D的样本的距离矩阵:
>>> from skbio import DistanceMatrix >>> dm = DistanceMatrix([[0., 0.21712454, 0.5007512, 0.91769271], ... [0.21712454, 0., 0.45995501, 0.80332382], ... [0.5007512, 0.45995501, 0., 0.65463348], ... [0.91769271, 0.80332382, 0.65463348, 0.]], ... ['A', 'B', 'C', 'D'])
中的每个样本定义元数据
pandas.DataFrame:>>> import pandas as pd >>> metadata = { ... 'A': {'body_site': 'skin'}, ... 'B': {'body_site': 'gut'}, ... 'C': {'body_site': 'gut'}, ... 'D': {'body_site': 'skin'}} >>> df = pd.DataFrame.from_dict(metadata, orient='index')
对距离矩阵运行主坐标分析(PCoA):
>>> from skbio.stats.ordination import pcoa >>> pcoa_results = pcoa(dm)
绘制排序结果图,其中每个样本按身体部位(分类变量)进行着色:
>>> fig = pcoa_results.plot(df=df, column='body_site', ... title='Samples colored by body site', ... cmap='Set1', s=50)
(
Source code,png)